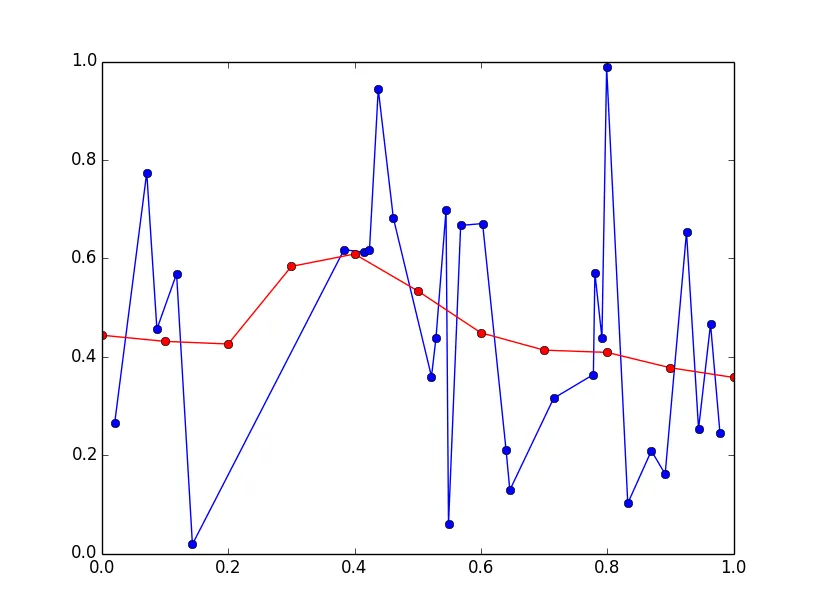
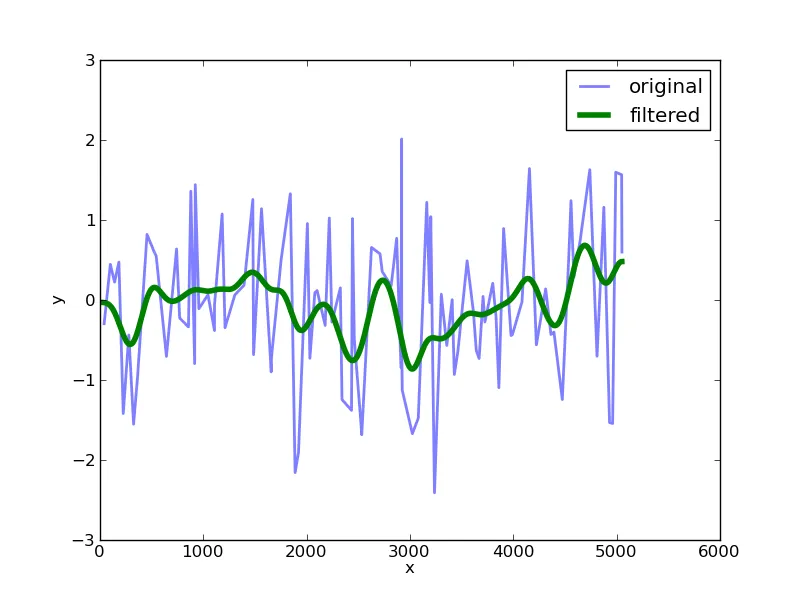
我有一组点(x,y),其中x和y是两个向量,例如:
from pylab import *
x = sorted(random(30))
y = random(30)
plot(x,y, 'o-')
现在我想用高斯函数对这个数据进行平滑处理,并仅在x轴上特定的(等间隔)点处进行评估。假设:
x_eval = linspace(0,1,11)
我得到的提示是这种方法叫做“高斯求和滤波器”,但是到目前为止我在numpy/scipy中没有找到任何实现,尽管乍一看它似乎是一个标准问题。 由于x值不是等间隔的,我不能使用scipy.ndimage.gaussian_filter1d。
通常,这种平滑处理是通过傅里叶空间进行的,并与核相乘,但我不知道是否可以用于不规则间距的数据。
感谢任何想法