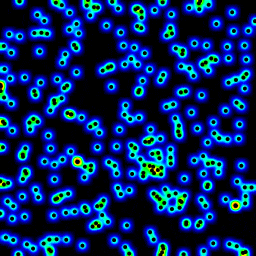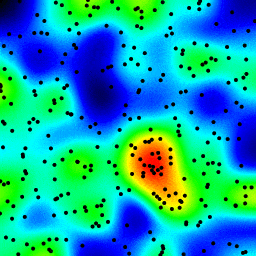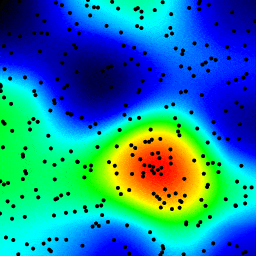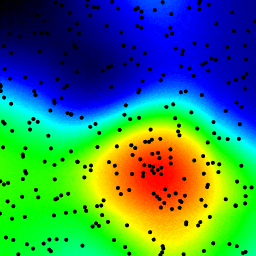
我查看了numpy、pyplot和scipy库,找到的最接近的是numpy.histogram2d。如下图所示,histogram2d的输出相当粗糙。(每个图像包含覆盖热力图的点,以便更好地理解)
第二次尝试是迭代所有数据点,然后根据距离计算热点值。这产生了一个外观更好的图像,但在我的应用程序中速度太慢。由于它是O(n),对于100个点可以正常工作,但使用我的实际数据集(30000个点)时会崩溃。
我的最后一次尝试是将数据存储在KDTree中,并使用最近的5个点来计算热点值。这个算法是O(1),因此对于大型数据集更快。它仍然不够快,生成一个256x256位图需要约20秒时间,我希望这个过程在大约1秒钟内完成。
编辑
6502提供的boxsum平滑解决方案在所有缩放级别下都很好,并且比我的原始方法快得多。
Luke和Neil G建议的高斯滤波器解决方案是最快的。
您可以在下面看到所有四种方法,总共使用1000个数据点,在3倍缩放时可见约60个点。
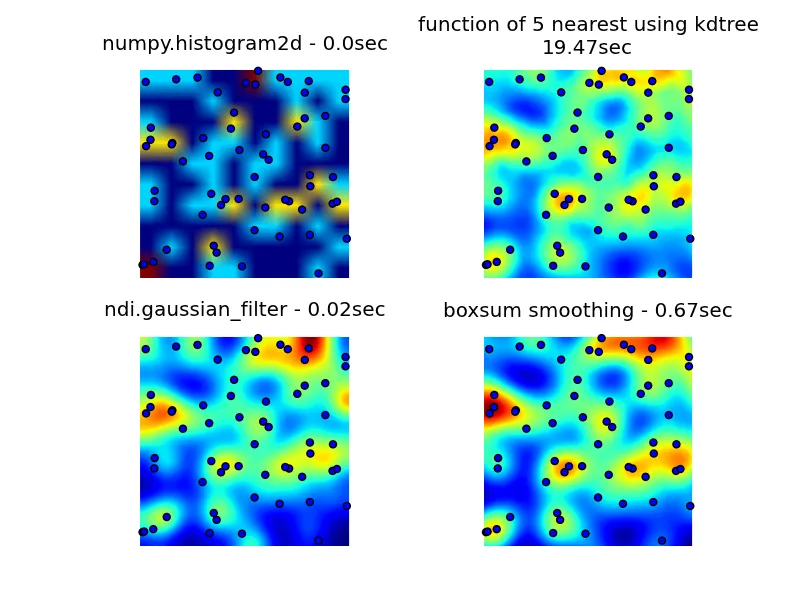
这里是生成我最初的3个尝试、由6502提供的boxsum平滑解决方案以及Luke建议的高斯滤波器(改进了处理边缘并允许放大)的完整代码:
import matplotlib
import numpy as np
from matplotlib.mlab import griddata
import matplotlib.cm as cm
import matplotlib.pyplot as plt
import math
from scipy.spatial import KDTree
import time
import scipy.ndimage as ndi
def grid_density_kdtree(xl, yl, xi, yi, dfactor):
zz = np.empty([len(xi),len(yi)], dtype=np.uint8)
zipped = zip(xl, yl)
kdtree = KDTree(zipped)
for xci in range(0, len(xi)):
xc = xi[xci]
for yci in range(0, len(yi)):
yc = yi[yci]
density = 0.
retvalset = kdtree.query((xc,yc), k=5)
for dist in retvalset[0]:
density = density + math.exp(-dfactor * pow(dist, 2)) / 5
zz[yci][xci] = min(density, 1.0) * 255
return zz
def grid_density(xl, yl, xi, yi):
ximin, ximax = min(xi), max(xi)
yimin, yimax = min(yi), max(yi)
xxi,yyi = np.meshgrid(xi,yi)
#zz = np.empty_like(xxi)
zz = np.empty([len(xi),len(yi)])
for xci in range(0, len(xi)):
xc = xi[xci]
for yci in range(0, len(yi)):
yc = yi[yci]
density = 0.
for i in range(0,len(xl)):
xd = math.fabs(xl[i] - xc)
yd = math.fabs(yl[i] - yc)
if xd < 1 and yd < 1:
dist = math.sqrt(math.pow(xd, 2) + math.pow(yd, 2))
density = density + math.exp(-5.0 * pow(dist, 2))
zz[yci][xci] = density
return zz
def boxsum(img, w, h, r):
st = [0] * (w+1) * (h+1)
for x in xrange(w):
st[x+1] = st[x] + img[x]
for y in xrange(h):
st[(y+1)*(w+1)] = st[y*(w+1)] + img[y*w]
for x in xrange(w):
st[(y+1)*(w+1)+(x+1)] = st[(y+1)*(w+1)+x] + st[y*(w+1)+(x+1)] - st[y*(w+1)+x] + img[y*w+x]
for y in xrange(h):
y0 = max(0, y - r)
y1 = min(h, y + r + 1)
for x in xrange(w):
x0 = max(0, x - r)
x1 = min(w, x + r + 1)
img[y*w+x] = st[y0*(w+1)+x0] + st[y1*(w+1)+x1] - st[y1*(w+1)+x0] - st[y0*(w+1)+x1]
def grid_density_boxsum(x0, y0, x1, y1, w, h, data):
kx = (w - 1) / (x1 - x0)
ky = (h - 1) / (y1 - y0)
r = 15
border = r * 2
imgw = (w + 2 * border)
imgh = (h + 2 * border)
img = [0] * (imgw * imgh)
for x, y in data:
ix = int((x - x0) * kx) + border
iy = int((y - y0) * ky) + border
if 0 <= ix < imgw and 0 <= iy < imgh:
img[iy * imgw + ix] += 1
for p in xrange(4):
boxsum(img, imgw, imgh, r)
a = np.array(img).reshape(imgh,imgw)
b = a[border:(border+h),border:(border+w)]
return b
def grid_density_gaussian_filter(x0, y0, x1, y1, w, h, data):
kx = (w - 1) / (x1 - x0)
ky = (h - 1) / (y1 - y0)
r = 20
border = r
imgw = (w + 2 * border)
imgh = (h + 2 * border)
img = np.zeros((imgh,imgw))
for x, y in data:
ix = int((x - x0) * kx) + border
iy = int((y - y0) * ky) + border
if 0 <= ix < imgw and 0 <= iy < imgh:
img[iy][ix] += 1
return ndi.gaussian_filter(img, (r,r)) ## gaussian convolution
def generate_graph():
n = 1000
# data points range
data_ymin = -2.
data_ymax = 2.
data_xmin = -2.
data_xmax = 2.
# view area range
view_ymin = -.5
view_ymax = .5
view_xmin = -.5
view_xmax = .5
# generate data
xl = np.random.uniform(data_xmin, data_xmax, n)
yl = np.random.uniform(data_ymin, data_ymax, n)
zl = np.random.uniform(0, 1, n)
# get visible data points
xlvis = []
ylvis = []
for i in range(0,len(xl)):
if view_xmin < xl[i] < view_xmax and view_ymin < yl[i] < view_ymax:
xlvis.append(xl[i])
ylvis.append(yl[i])
fig = plt.figure()
# plot histogram
plt1 = fig.add_subplot(221)
plt1.set_axis_off()
t0 = time.clock()
zd, xe, ye = np.histogram2d(yl, xl, bins=10, range=[[view_ymin, view_ymax],[view_xmin, view_xmax]], normed=True)
plt.title('numpy.histogram2d - '+str(time.clock()-t0)+"sec")
plt.imshow(zd, origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
# plot density calculated with kdtree
plt2 = fig.add_subplot(222)
plt2.set_axis_off()
xi = np.linspace(view_xmin, view_xmax, 256)
yi = np.linspace(view_ymin, view_ymax, 256)
t0 = time.clock()
zd = grid_density_kdtree(xl, yl, xi, yi, 70)
plt.title('function of 5 nearest using kdtree\n'+str(time.clock()-t0)+"sec")
cmap=cm.jet
A = (cmap(zd/256.0)*255).astype(np.uint8)
#A[:,:,3] = zd
plt.imshow(A , origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
# gaussian filter
plt3 = fig.add_subplot(223)
plt3.set_axis_off()
t0 = time.clock()
zd = grid_density_gaussian_filter(view_xmin, view_ymin, view_xmax, view_ymax, 256, 256, zip(xl, yl))
plt.title('ndi.gaussian_filter - '+str(time.clock()-t0)+"sec")
plt.imshow(zd , origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
# boxsum smoothing
plt3 = fig.add_subplot(224)
plt3.set_axis_off()
t0 = time.clock()
zd = grid_density_boxsum(view_xmin, view_ymin, view_xmax, view_ymax, 256, 256, zip(xl, yl))
plt.title('boxsum smoothing - '+str(time.clock()-t0)+"sec")
plt.imshow(zd, origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
if __name__=='__main__':
generate_graph()
plt.show()
for循环迭代。通过使用numpy重写代码后,我加速了100倍。例如:for i in np.arange(1e5): x[i] =+ 1应该比x =+ 1慢,因为后者是numpy,而前者并没有真正利用numpy的加速优势。 - ahelm