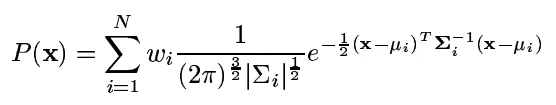 其中N个不同的核具有指定权重w、均值和对角协方差矩阵。
其中N个不同的核具有指定权重w、均值和对角协方差矩阵。所以基本上我想要一个函数
compute_densities(image, kernels) -> numpy array of floats。在Python中,最有效的方法是什么?如果scipy中没有库函数进行此操作,我会感到惊讶,但我很久以前就学过统计学,所以我有点困惑于文档的细节。基本上我想要以下内容,只是比naive python更高效(2pi^{-3/2}被忽略,因为它是对我来说无关紧要的常数因子,我只关心概率之间的比率)。
def compute_probabilities(img, kernels):
np.seterr(divide='ignore') # 1 / covariance logs an error otherwise
result = np.zeros((img.shape[0], img.shape[1]))
for row_pos, row_val in enumerate(img):
for col_pos, val in enumerate(row_val):
prob = 0.0
for kernel in kernels:
mean, covariance, weight = kernel
val_sub_mu = np.array([val]).T - mean
cov_inv = np.where(covariance != 0, 1 / covariance, 0)
tmp = val_sub_mu.T.dot(cov_inv).dot(val_sub_mu)
prob += weight / np.sqrt(np.linalg.norm(covariance)) * \
math.exp(-0.5 * tmp)
result[row_pos][col_pos] = prob
np.seterr(divide='warn')
return result
输入: 用cv2.imread打开某个jpg文件,它会返回一个包含三个颜色通道的三元素uint8结构的二维数组(高x宽)。
Kernels是一个namedtuple('Kernel', 'mean covariance weight'),其中mean是一个向量,covariance是一个除对角线外其余元素均为零的3x3矩阵,而weight是一个浮点数,0 < weight < 1。为简单起见,只需指定对角线,然后在转换为3x3矩阵时即可(表示方法不一定固定,可以自由更改所有内容)。
some_kernels = [
Kernel(np.array([(73.53, 29.94, 17.76)]), np.array([(765.40, 121.44, 112.80)]), 0.0294),
...
]
def fixup_kernels(kernels):
new_kernels = []
for kernel in kernels:
cov = np.zeros((3, 3))
for pos, c in enumerate(kernel.covariance[0]):
cov[pos][pos] = c
new_kernels.append(Kernel(kernel.mean.T, cov, kernel.weight))
return new_kernels
some_kernels = fixup_kernels(some_kernels)
img = cv2.imread("something.jpg")
result = compute_probabalities(img, some_kernels)