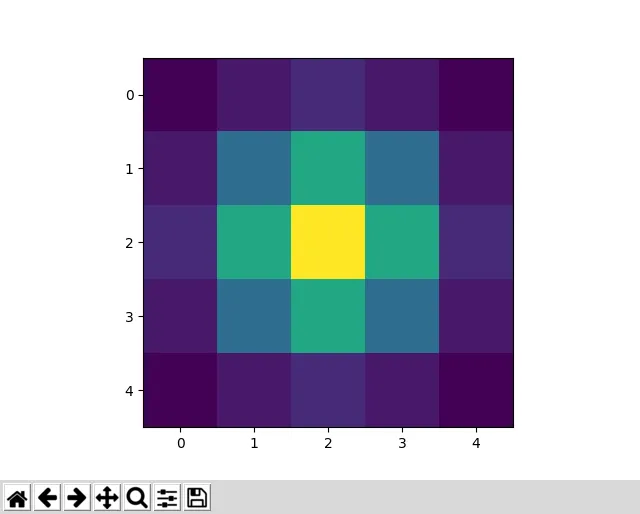
我希望获得一个大小为m行和n列的高斯窗口。
我知道如何得到一维的高斯窗口,即下面这样。
from scipy.stats import multivariate_normal
multivariate_normal(mean=[1, 5], cov=(2.5))
现在我需要一个矩阵的两个维度。 目的:我想将这个滤镜放在图片的顶部。绿色是一张图片的矩阵。蓝色圆圈是高斯滤镜。我不确定如何得到蓝色窗口。
我考虑应用类似于以下内容的某些东西 -
gw = multivariate_normal(mean=[1, 5], cov=(2.5))
for i in range(image.shape[0):
image_gauss_window[i:] = gw
你能提供一种找出图像高斯滤波器的方法吗?我看到OpenCV中有很多函数将高斯模糊应用于图像。但是这里我想要在将其应用/卷积到图像之前找到过滤器。

size-1而不是直接使用size? - information_interchange