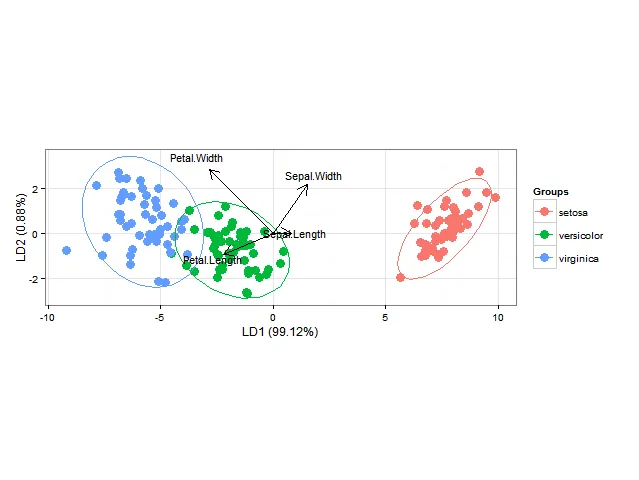
使用ggord可以制作漂亮的线性判别分析ggplot2双标图(参见M. Greenacre的“实践中的双标图”第11章,图11.5),如下所示:
library(MASS)
install.packages("devtools")
library(devtools)
install_github("fawda123/ggord")
library(ggord)
data(iris)
ord <- lda(Species ~ ., iris, prior = rep(1, 3)/3)
ggord(ord, iris$Species)
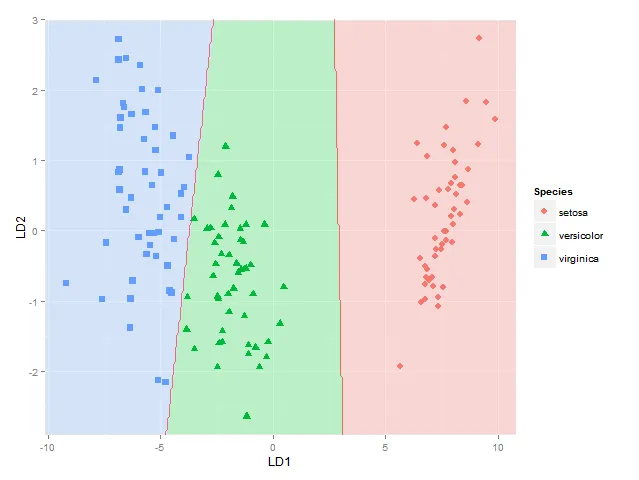
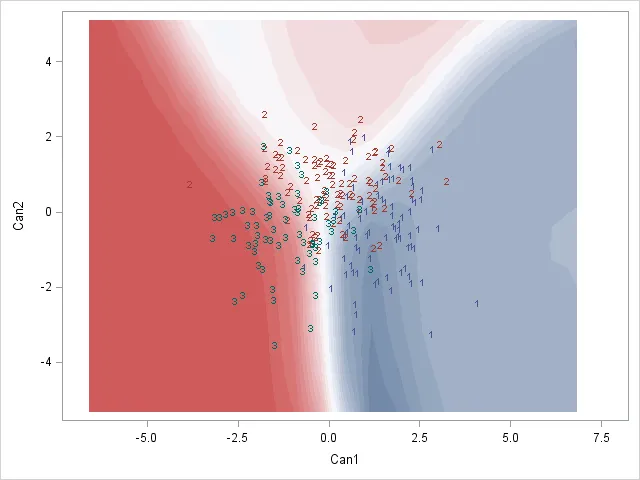
我还想添加分类区域(用与各自组相同颜色的实心区域表示,alpha=0.5)或类成员后验概率(然后根据此后验概率和用于每个组的相同颜色变化alpha),(就像在 BiplotGUI 中所做的那样,但我正在寻找一种 ggplot2 的解决方案)。有谁知道如何使用 ggplot2 做到这一点,也许使用geom_tile?
编辑:下面有人问如何计算后验分类概率和预测类别。方法如下:
library(MASS)
library(ggplot2)
library(scales)
fit <- lda(Species ~ ., data = iris, prior = rep(1, 3)/3)
datPred <- data.frame(Species=predict(fit)$class,predict(fit)$x)
#Create decision boundaries
fit2 <- lda(Species ~ LD1 + LD2, data=datPred, prior = rep(1, 3)/3)
ld1lim <- expand_range(c(min(datPred$LD1),max(datPred$LD1)),mul=0.05)
ld2lim <- expand_range(c(min(datPred$LD2),max(datPred$LD2)),mul=0.05)
ld1 <- seq(ld1lim[[1]], ld1lim[[2]], length.out=300)
ld2 <- seq(ld2lim[[1]], ld1lim[[2]], length.out=300)
newdat <- expand.grid(list(LD1=ld1,LD2=ld2))
preds <-predict(fit2,newdata=newdat)
predclass <- preds$class
postprob <- preds$posterior
df <- data.frame(x=newdat$LD1, y=newdat$LD2, class=predclass)
df$classnum <- as.numeric(df$class)
df <- cbind(df,postprob)
head(df)
x y class classnum setosa versicolor virginica
1 -10.122541 -2.91246 virginica 3 5.417906e-66 1.805470e-10 1
2 -10.052563 -2.91246 virginica 3 1.428691e-65 2.418658e-10 1
3 -9.982585 -2.91246 virginica 3 3.767428e-65 3.240102e-10 1
4 -9.912606 -2.91246 virginica 3 9.934630e-65 4.340531e-10 1
5 -9.842628 -2.91246 virginica 3 2.619741e-64 5.814697e-10 1
6 -9.772650 -2.91246 virginica 3 6.908204e-64 7.789531e-10 1
colorfun <- function(n,l=65,c=100) { hues = seq(15, 375, length=n+1); hcl(h=hues, l=l, c=c)[1:n] } # default ggplot2 colours
colors <- colorfun(3)
colorslight <- colorfun(3,l=90,c=50)
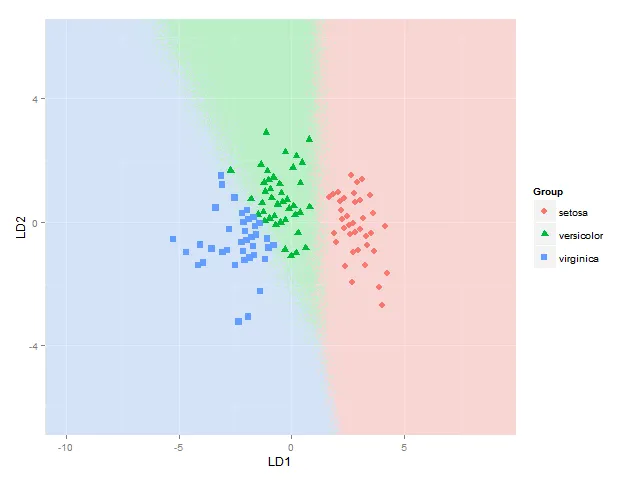
ggplot(datPred, aes(x=LD1, y=LD2) ) +
geom_raster(data=df, aes(x=x, y=y, fill = factor(class)),alpha=0.7,show_guide=FALSE) +
geom_contour(data=df, aes(x=x, y=y, z=classnum), colour="red2", alpha=0.5, breaks=c(1.5,2.5)) +
geom_point(data = datPred, size = 3, aes(pch = Species, colour=Species)) +
scale_x_continuous(limits = ld1lim, expand=c(0,0)) +
scale_y_continuous(limits = ld2lim, expand=c(0,0)) +
scale_fill_manual(values=colorslight,guide=F)
虽然用等高线/间断点在1.5和2.5处显示分类边界的方法并不总是正确的——它对于物种1和2以及物种2和3之间的边界是正确的,但如果物种1的区域与物种3相邻,我会得到两个边界——也许我需要使用这里使用的方法,单独考虑每个物种对之间的边界)
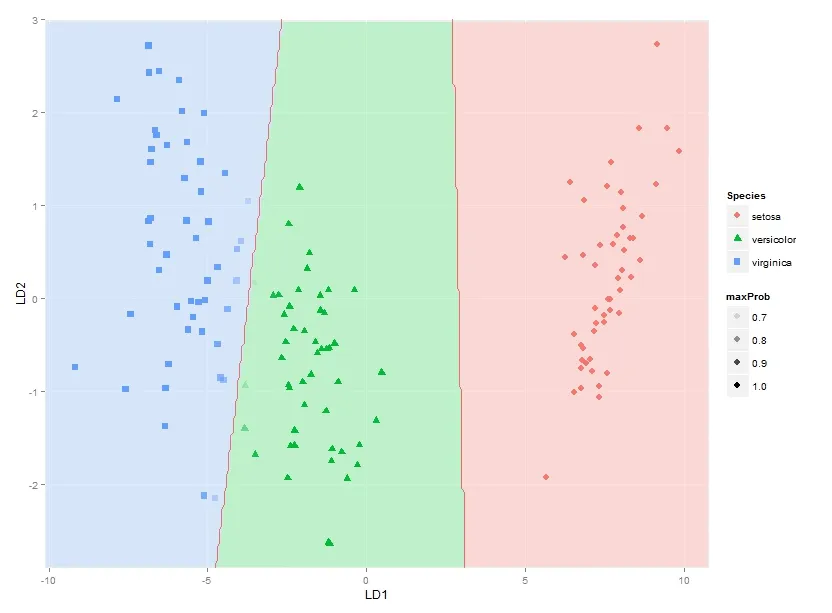
这让我完成了分类区域的绘制。我还在寻找一种解决方案,可以在每个坐标上绘制每个物种的后验分类概率,使用alpha(不透明度)与每个物种的后验分类概率成比例,以及一个特定于物种的颜色。换句话说,用三张叠加的图像堆栈。由于ggplot2中的混合 alpha 是已知的依赖顺序的,我认为这个堆栈的颜色必须预先计算,然后使用类似于以下内容的方法绘制。
qplot(x, y, data=mydata, fill=rgb, geom="raster") + scale_fill_identity()
也许有人知道如何做到这一点吗?或者是否有任何想法以最佳方式表示这些后验分类概率?
请注意,此方法应适用于任意数量的组,而不仅仅适用于此特定示例。
expand_limits()而不是expand_range()? - lgautier