如果我理解你的问题正确,你的输入“观测”数据是定期网格化的吗?
如果是这样,
scipy.ndimage.map_coordinates 正是你想要的。
一开始有点难理解,但本质上,你只需要提供一个坐标序列,以像素/体素/n维索引坐标的形式插值网格值。
以2D为例:
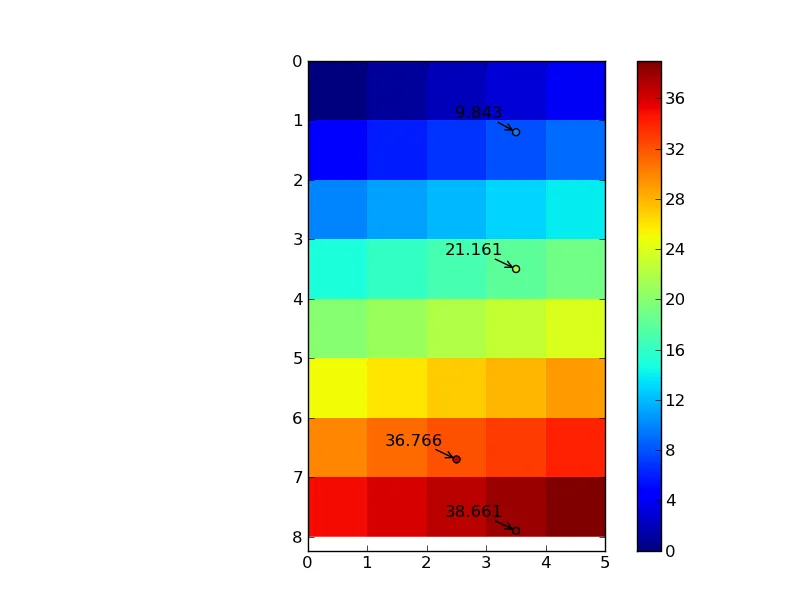
import numpy as np
from scipy import ndimage
import matplotlib.pyplot as plt
data = np.arange(40).reshape((8,5)).astype(np.float)
coords = np.array([[1.2, 3.5], [6.7, 2.5], [7.9, 3.5], [3.5, 3.5]])
coords = coords.T
zi = ndimage.map_coordinates(data, coords, order=3, mode='nearest')
row, column = coords
nrows, ncols = data.shape
im = plt.imshow(data, interpolation='nearest', extent=[0, ncols, nrows, 0])
plt.colorbar(im)
plt.scatter(column, row, c=zi, vmin=data.min(), vmax=data.max())
for r, c, z in zip(row, column, zi):
plt.annotate('%0.3f' % z, (c,r), xytext=(-10,10), textcoords='offset points',
arrowprops=dict(arrowstyle='->'), ha='right')
plt.show()
要在n维中完成这个操作,我们只需要传入相应大小的数组:
import numpy as np
from scipy import ndimage
data = np.arange(3*5*9).reshape((3,5,9)).astype(np.float)
coords = np.array([[1.2, 3.5, 7.8], [0.5, 0.5, 6.8]])
zi = ndimage.map_coordinates(data, coords.T)
就缩放和内存使用而言,如果您使用的是顺序> 1(即非线性插值),
map_coordinates将创建一个过滤后的数组副本。 如果您只想在很少的点上进行插值,则这是一个相当大的开销。 但是,它不会随着您想要插值的点数增加而增加。 只要有足够的RAM来存储输入数据数组的单个临时副本,就可以了。
如果您无法在内存中存储数据副本,则可以a)指定
prefilter = False和
order = 1并使用线性插值,或者b)使用
ndimage.spline_filter替换原始数据的过滤版本,然后使用
prefilter = False调用map_coordinates。
即使您有足够的内存,如果需要多次调用map_coordinates(例如交互式使用等),保留过滤后的数据集也可以大大加快速度。
