我试图按照这个示例进行操作,但似乎无法适应我的数据集,因为我需要截断正态分布:
https://dev59.com/mVsV5IYBdhLWcg3wpQF8=
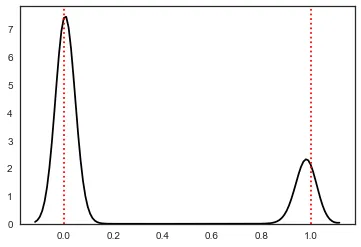
我的数据集肯定是两个被截断的正态分布的混合物。域中的最小值为0,最大值为1。我想创建一个对象,以优化参数并获取从该分布中绘制一系列数字的可能性。一种选择可能是只使用KDE模型,并使用pdf来获取可能性。然而,我想要两个分布的确切均值和标准差。我猜我可以将数据分成两半,然后分别对两个正态分布进行建模,但我也想学习如何在SciPy中使用optimize。我刚开始尝试这种统计分析,如果这看起来很幼稚,请谅解。
我不确定如何以这种方式获得一个pdf,它可以积分为1,并且其定义域被限制在0到1之间。
我不确定如何以这种方式获得一个pdf,它可以积分为1,并且其定义域被限制在0到1之间。
import requests
from ast import literal_eval
from scipy import optimize, stats
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
# Actual Data
u = np.asarray(literal_eval(requests.get("https://pastebin.com/raw/hP5VJ9vr").text))
# u.size ==> 6000
u.min(), u.max()
# (1.3628525454666037e-08, 0.99973136607553781)
# Distribution
with plt.style.context("seaborn-white"):
fig, ax = plt.subplots()
sns.kdeplot(u, color="black", ax=ax)
ax.axvline(0, linestyle=":", color="red")
ax.axvline(1, linestyle=":", color="red")
kde = stats.gaussian_kde(u)
# KDE Model
def truncated_gaussian_lower(x,mu,sigma,A):
return np.clip(A*np.exp(-(x-mu)**2/2/sigma**2), a_min=0, a_max=None)
def truncated_gaussian_upper(x,mu,sigma,A):
return np.clip(A*np.exp(-(x-mu)**2/2/sigma**2), a_min=None, a_max=1)
def mixture_model(x,mu1,sigma1,A1,mu2,sigma2,A2):
return truncated_gaussian_lower(x,mu1,sigma1,A1) + truncated_gaussian_upper(x,mu2,sigma2,A2)
kde = stats.gaussian_kde(u)
# Estimates: mu sigma A
estimates= [0.1, 1, 3,
0.9, 1, 1]
params,cov= optimize.curve_fit(mixture_model,u,kde.pdf(u),estimates )
# ---------------------------------------------------------------------------
# RuntimeError Traceback (most recent call last)
# <ipython-input-265-b2efb2ca0e0a> in <module>()
# 32 estimates= [0.1, 1, 3,
# 33 0.9, 1, 1]
# ---> 34 params,cov= optimize.curve_fit(mixture_model,u,kde.pdf(u),estimates )
# /Users/mu/anaconda/lib/python3.6/site-packages/scipy/optimize/minpack.py in curve_fit(f, xdata, ydata, p0, sigma, absolute_sigma, check_finite, bounds, method, jac, **kwargs)
# 738 cost = np.sum(infodict['fvec'] ** 2)
# 739 if ier not in [1, 2, 3, 4]:
# --> 740 raise RuntimeError("Optimal parameters not found: " + errmsg)
# 741 else:
# 742 # Rename maxfev (leastsq) to max_nfev (least_squares), if specified.
# RuntimeError: Optimal parameters not found: Number of calls to function has reached maxfev = 1400.
回应@Uvar下面非常有帮助的解释。我正在尝试测试从0到1的积分是否等于1,但我得到的是0.3。我认为我的逻辑中缺少了一个关键步骤:
# KDE Model
def truncated_gaussian(x,mu,sigma,A):
return A*np.exp(-(x-mu)**2/2/sigma**2)
def mixture_model(x,mu1,sigma1,A1,mu2,sigma2,A2):
if type(x) == np.ndarray:
norm_probas = truncated_gaussian(x,mu1,sigma1,A1) + truncated_gaussian(x,mu2,sigma2,A2)
mask_lower = x < 0
mask_upper = x > 1
mask_floor = (mask_lower.astype(int) + mask_upper.astype(int)) > 1
norm_probas[mask_floor] = 0
return norm_probas
else:
if (x < 0) or (x > 1):
return 0
return truncated_gaussian_lower(x,mu1,sigma1,A1) + truncated_gaussian_upper(x,mu2,sigma2,A2)
kde = stats.gaussian_kde(u, bw_method=2e-2)
# # Estimates: mu sigma A
estimates= [0.1, 1, 3,
0.9, 1, 1]
params,cov= optimize.curve_fit(mixture_model,u,kde.pdf(u)/integrate.quad(kde, 0 , 1)[0],estimates ,maxfev=5000)
# params
# array([ 9.89751700e-01, 1.92831695e-02, 7.84324114e+00,
# 3.73623345e-03, 1.07754038e-02, 3.79238972e+01])
# Test the integral from 0 - 1
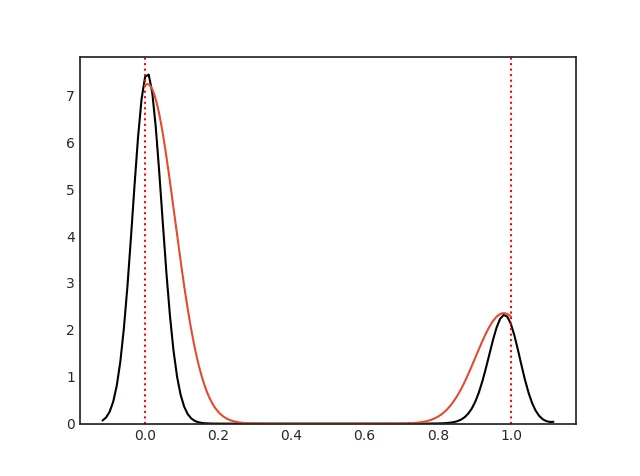
x = np.linspace(0,1,1000)
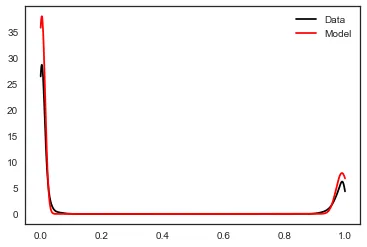
with plt.style.context("seaborn-white"):
fig, ax = plt.subplots()
ax.plot(x, kde(x), color="black", label="Data")
ax.plot(x, mixture_model(x, *params), color="red", label="Model")
ax.legend()
# Integrating from 0 to 1
integrate.quad(lambda x: mixture_model(x, *params), 0,1)[0]
# 0.3026863969781809