我正在尝试使用SciPy Skewnorm包将数据拟合成偏态正态分布。
然而,我无法正确理解用法,因为我找不到关于这个问题的适当文档或示例。
在帮助部分中,我找到了文档,并尝试使用
如果有人能帮忙,请告诉我。
然而,我无法正确理解用法,因为我找不到关于这个问题的适当文档或示例。
在帮助部分中,我找到了文档,并尝试使用
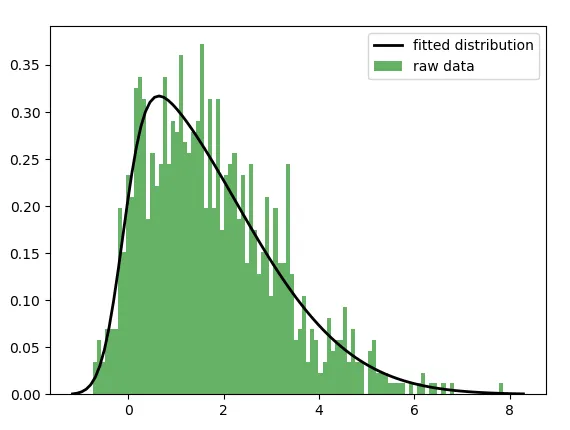
skewnorm.fit()和skewnorm.pdf()来将数据拟合成模型,并使用该模型输出分布并与原始数据进行比较。如果有人能帮忙,请告诉我。
from scipy import stats
import matplotlib.pyplot as plt
import numpy as np
# choose some parameters
a, loc, scale = 5.3, -0.1, 2.2
# draw a sample
data = stats.skewnorm(a, loc, scale).rvs(1000)
# estimate parameters from sample
ae, loce, scalee = stats.skewnorm.fit(data)
# Plot the PDF.
plt.figure()
plt.hist(data, bins=100, normed=True, alpha=0.6, color='g')
xmin, xmax = plt.xlim()
x = np.linspace(xmin, xmax, 100)
p = stats.skewnorm.pdf(x,ae, loce, scalee)#.rvs(100)
plt.plot(x, p, 'k', linewidth=2)
输出:
fit()方法,那么您将数据建模为从偏斜正态分布中随机抽取的样本,并且您想要估计该分布的参数。是这样吗?如果是这样,您是否尝试过像params = skewnorm.fit(data)这样简单的东西,其中data是包含输入值的1-d数组或序列?(请参见Paul Panzer的答案示例。) - Warren Weckesser