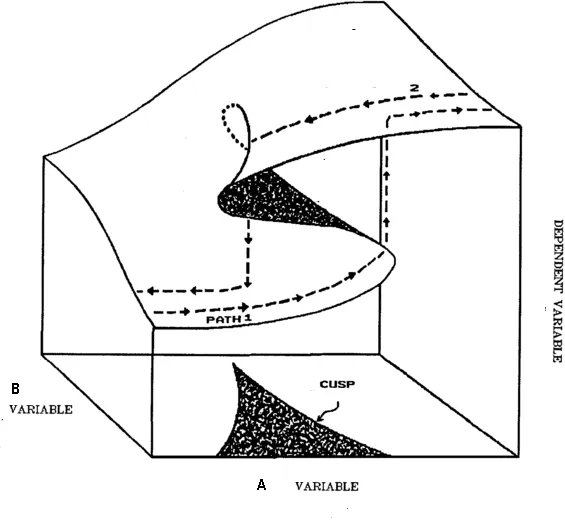
我正在尝试绘制一个分叉齿形随时间演变的图。 x 和y之间的关系一开始大约是线性的,但最后变成了S形状。最终的关系不是函数;对于某些x值,有多个y值。
Matplotlib可以为曲面图绘制不错的线框图,但这些曲面图似乎不能处理非函数。
是否有另一种方法只绘制此关系的表面?(如果可能,我不想要一个实心的形状。)
目前,我的数据在零数组中,其中 1 表示表面位置的近似值。
我已经包含了一个非常小的样本数据集和将绘制其位置的样本代码。如何“连接这些点”?
我的实际数据集更大(500x200x200)且各不相同,因此我需要开发一个灵活的系统。
这就是最终的图像可能会看起来像:
从这里阅读文档来看,似乎我需要将我的数据转换为2D数组。 如果是这种情况,请提供一种方法,并尽可能告诉我这些数组代表什么。
非常感谢任何推进此事的评论/建议。
import numpy as np
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
sample_data = np.array([
[[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 1., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 1., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 1., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 1., 1.]],
[[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 1., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 1., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 1., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 1., 1.]],
[[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 1., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 1., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 1., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 1., 1.]],
[[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 1., 1., 1., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 1., 1., 1., 1., 1.]],
[[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 1., 1., 1., 1., 1., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 1., 1., 1., 1., 1., 1., 1., 1.]],
[[ 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 1., 1., 1., 1., 1., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 1., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 0., 0., 1., 0., 0., 0.],
[ 0., 0., 0., 0., 0., 1., 1., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 1., 0., 0., 0., 0., 0., 0.],
[ 0., 0., 0., 1., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 1., 1., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 1., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.],
[ 0., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.]]
] )
XS, YS, ZS = [],[],[]
for g in xrange(np.shape(sample_data)[0]):
for row in xrange(np.shape(sample_data)[1]):
for col in xrange(np.shape(sample_data)[2]):
if sample_data[g][row][col] == 1:
XS.append(g)
YS.append(col)
ZS.append(row)
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
ax.scatter(XS, YS, ZS)
plt.show()
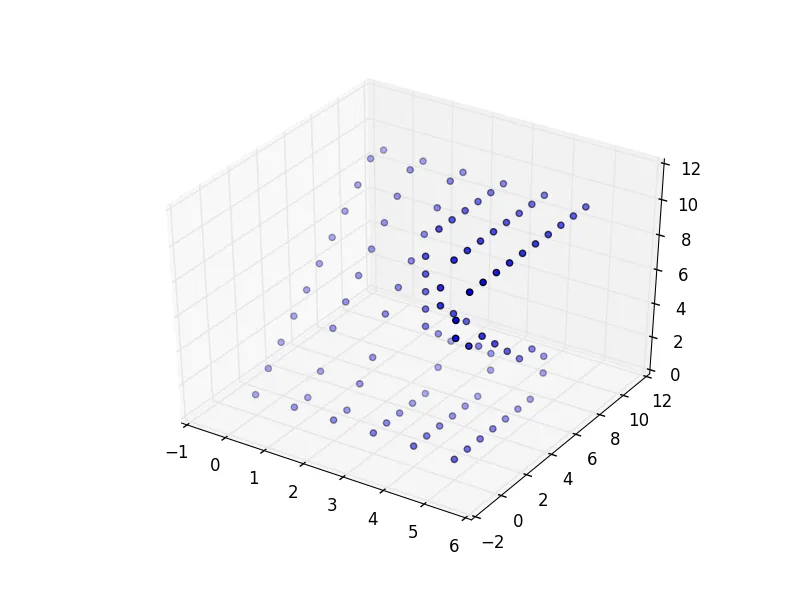
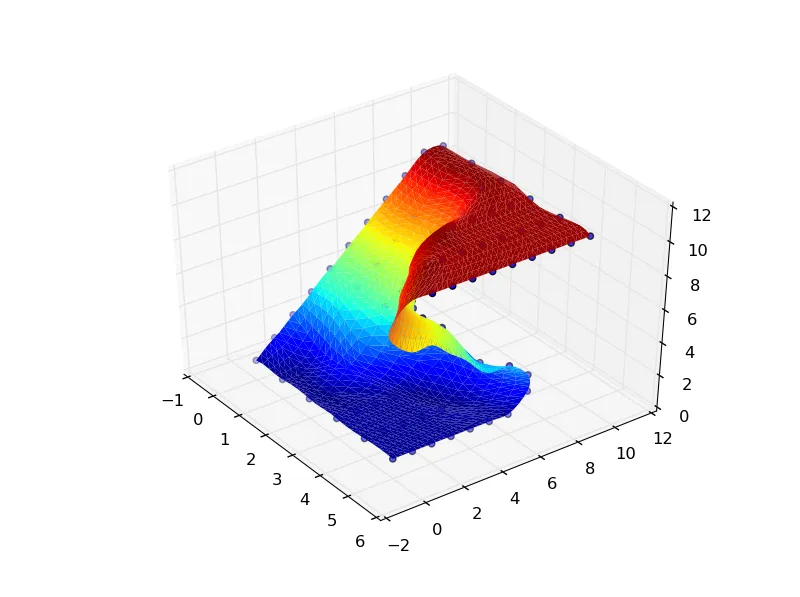 以下是基于您下面的代码的示例(由于您的数据非常粗糙,我添加了一些插值):
以下是基于您下面的代码的示例(由于您的数据非常粗糙,我添加了一些插值):