在Python中进行加权高斯核密度估计
15
- Till Hoffmann
1
2截至scipy版本1.2,sp.stats.gaussian_kde中似乎有一个“weights”选项,它似乎可以实现您现在想要的功能。 - wmsmith
3个回答
26
sklearn.neighbors.KernelDensity和 statsmodels.nonparametric 都似乎不支持加权样本。我修改了 scipy.stats.gaussian_kde 以允许异构采样权重,并认为这些结果可能对其他人有用。下面是一个示例。
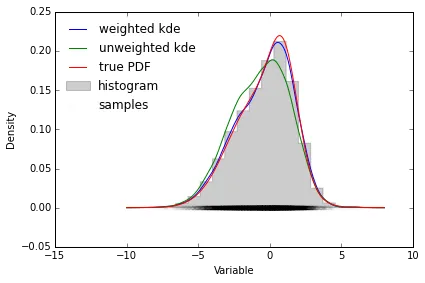
ipython笔记本可以在此处找到:http://nbviewer.ipython.org/gist/tillahoffmann/f844bce2ec264c1c8cb5
实现细节
加权算术平均值为
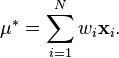
然后给出无偏数据协方差矩阵:
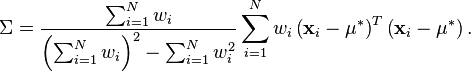
带宽可以使用 scipy 中的 scott 或 silverman 规则选择。但是,用于计算带宽的样本数量是 Kish 的有效样本大小的近似公式。
- Till Hoffmann
10
2你是否考虑过向
scipy 或 statsmodels 的开发人员提出请求,将你的代码集成到这些库中? - cel3是的,但我还没有开始实现重采样和积分。一旦完成,我将发起一个拉取请求。 - Till Hoffmann
我一直在解决类似的问题,但是使用自己的框架而不是修改scipy。我没有想到使用Kish的近似方法。你认为这是最好的带宽估计器吗?它会重新加权数据集中的每个点,使其具有相同的有效样本大小。我想知道是否使用可变带宽可能更合理。 - Gabriel
没问题,但是最好使用
np.arange而不是xrange,因为numpy经常在内部转换为数组。但是测试比我的猜测更好。 - Till Hoffmann请注意,正如@Ramon在下面指出的那样,
statsmodels.nonparametric.kde.KDEUnivariate现在支持weights。 - Lei显示剩余5条评论
2
针对一元分布,您可以使用statsmodels中的
这会生成以下图像: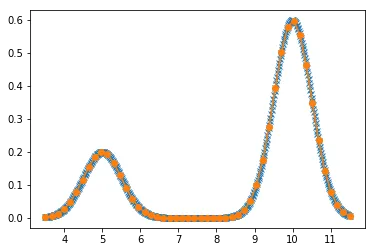
KDEUnivariate。它的文档不是很全面,但是fit方法接受weights参数。这时您不能使用FFT。以下是一个示例:import matplotlib.pyplot as plt
from statsmodels.nonparametric.kde import KDEUnivariate
kde1= KDEUnivariate(np.array([10.,10.,10.,5.]))
kde1.fit(bw=0.5)
plt.plot(kde1.support, [kde1.evaluate(xi) for xi in kde1.support],'x-')
kde1= KDEUnivariate(np.array([10.,5.]))
kde1.fit(weights=np.array([3.,1.]),
bw=0.5,
fft=False)
plt.plot(kde1.support, [kde1.evaluate(xi) for xi in kde1.support], 'o-')
这会生成以下图像:

- Ramon Crehuet
1
请查看PyQT-Fit和Python的统计包。它们似乎具有使用加权观测值的核密度估计功能。
- dikdirk
1
请注意,从1.3.4版本开始,PyQT-Fit仅支持一维核密度估计。 - lapis
网页内容由stack overflow 提供, 点击上面的可以查看英文原文,
原文链接
原文链接