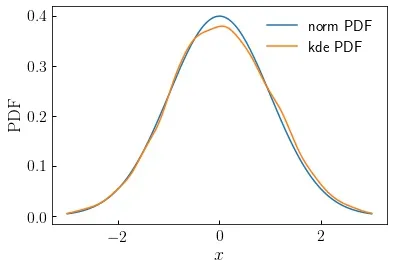
scipy.stats中的gaussian_kde函数具有一个evaluate函数,可以返回输入点的概率密度函数(PDF)的值。我正在尝试使用gaussian_kde来估计反向CDF。这样做的动机是为了生成一些输入数据的蒙特卡罗实现,其统计分布是使用KDE进行数值估计的。是否有一种与gaussian_kde相关联的方法可以用于此目的?下面的示例演示了如何处理高斯分布的情况。首先,我展示了如何进行PDF计算以设置我要实现的具体API:
import numpy as np
from scipy.stats import norm, gaussian_kde
npts_kde = int(5e3)
n = np.random.normal(loc=0, scale=1, size=npts_kde)
kde = gaussian_kde(n)
npts_sample = int(1e3)
x = np.linspace(-3, 3, npts_sample)
kde_pdf = kde.evaluate(x)
norm_pdf = norm.pdf(x)
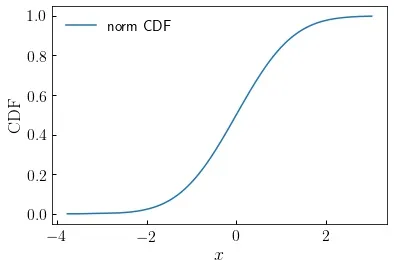
是否有类似简单的方式来计算逆 CDF?norm 函数具有非常方便的 isf 函数,可以完全做到这一点:
cdf_value = np.sort(np.random.rand(npts_sample))
cdf_inv = norm.isf(1 - cdf_value)
kde_gaussian 是否存在这样的函数?或者是否可以通过已经实现的方法简单构造出这样的函数?