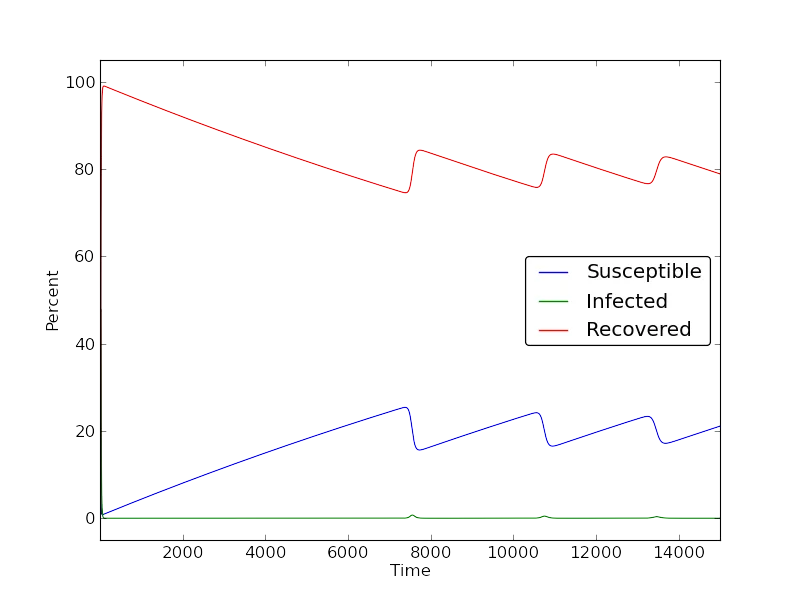
我正在实现一个非常简单的易感-感染-恢复模型,该模型针对一个稳定人口进行建模,这是一个相当琐碎的任务。但是,我在使用PysCeS或SciPy时遇到了求解器错误,它们都使用lsoda作为其基础求解器。这仅发生在特定参数值下,我对此感到困惑。我使用的代码如下:
import numpy as np
from pylab import *
import scipy.integrate as spi
#Parameter Values
S0 = 99.
I0 = 1.
R0 = 0.
PopIn= (S0, I0, R0)
beta= 0.50
gamma=1/10.
mu = 1/25550.
t_end = 15000.
t_start = 1.
t_step = 1.
t_interval = np.arange(t_start, t_end, t_step)
#Solving the differential equation. Solves over t for initial conditions PopIn
def eq_system(PopIn,t):
'''Defining SIR System of Equations'''
#Creating an array of equations
Eqs= np.zeros((3))
Eqs[0]= -beta * (PopIn[0]*PopIn[1]/(PopIn[0]+PopIn[1]+PopIn[2])) - mu*PopIn[0] + mu*(PopIn[0]+PopIn[1]+PopIn[2])
Eqs[1]= (beta * (PopIn[0]*PopIn[1]/(PopIn[0]+PopIn[1]+PopIn[2])) - gamma*PopIn[1] - mu*PopIn[1])
Eqs[2]= gamma*PopIn[1] - mu*PopIn[2]
return Eqs
SIR = spi.odeint(eq_system, PopIn, t_interval)
这会产生以下错误:
lsoda-- at current t (=r1), mxstep (=i1) steps
taken on this call before reaching tout
In above message, I1 = 500
In above message, R1 = 0.7818108252072E+04
Excess work done on this call (perhaps wrong Dfun type).
Run with full_output = 1 to get quantitative information.
通常当我遇到这样的问题时,我设置的方程系统中会有一些终端错误,但我却看不出任何问题。奇怪的是,如果将μ更改为类似于1/15550的东西,它也可以工作。以防系统出现问题,我还在R中实现了如下模型:
require(deSolve)
sir.model <- function (t, x, params) {
S <- x[1]
I <- x[2]
R <- x[3]
with (
as.list(params),
{
dS <- -beta*S*I/(S+I+R) - mu*S + mu*(S+I+R)
dI <- beta*S*I/(S+I+R) - gamma*I - mu*I
dR <- gamma*I - mu*R
res <- c(dS,dI,dR)
list(res)
}
)
}
times <- seq(0,15000,by=1)
params <- c(
beta <- 0.50,
gamma <- 1/10,
mu <- 1/25550
)
xstart <- c(S = 99, I = 1, R= 0)
out <- as.data.frame(lsoda(xstart,times,sir.model,params))
这个也使用了 lsoda,但似乎一切顺利。有人能看出 Python 代码出了什么问题吗?