我正在尝试拟合通过CCD上的光谱仪检测到的线轮廓。为了方便考虑,我包含了一个演示,如果解决了,与我实际想要解决的问题非常相似。
我已经查看了这个链接:https://stats.stackexchange.com/questions/46626/fitting-model-for-two-normal-distributions-in-pymc和其他各种问题和答案,但它们所做的事情与我想做的根本不同。
请注意,我实际要拟合的函数并不是高斯函数——因此,请使用通用函数(例如我的示例中的GaussFunc),而不是“内置”的pymc.Normal()类型函数来提供示例。
此外,我了解模型选择是另一个问题:因此,在当前噪声下,可能只有1个分量(轮廓)是统计上证明的。但我想看看1、2、3等分量的最佳解决方案。
我也不固执于使用PyMC——如果scikit-learn、astroML或其他某个包似乎完美,请告诉我!
编辑:
但我无法构建一个可行的mc.model。
我已经查看了这个链接:https://stats.stackexchange.com/questions/46626/fitting-model-for-two-normal-distributions-in-pymc和其他各种问题和答案,但它们所做的事情与我想做的根本不同。
import pymc as mc
import numpy as np
import pylab as pl
def GaussFunc(x, amplitude, centroid, sigma):
return amplitude * np.exp(-0.5 * ((x - centroid) / sigma)**2)
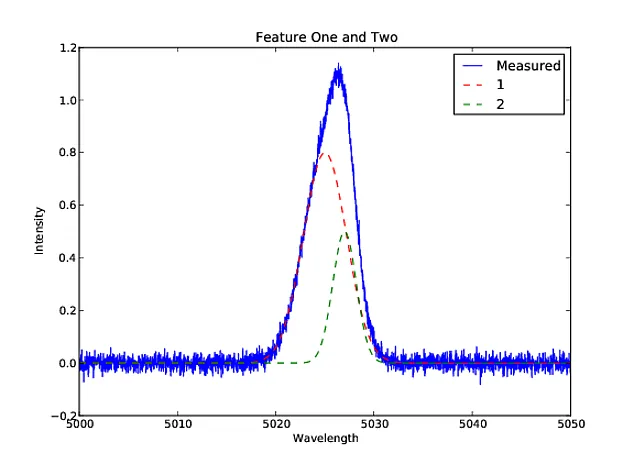
wavelength = np.arange(5000, 5050, 0.02)
# Profile 1
centroid_one = 5025.0
sigma_one = 2.2
height_one = 0.8
profile1 = GaussFunc(wavelength, height_one, centroid_one, sigma_one, )
# Profile 2
centroid_two = 5027.0
sigma_two = 1.2
height_two = 0.5
profile2 = GaussFunc(wavelength, height_two, centroid_two, sigma_two, )
# Measured values
noise = np.random.normal(0.0, 0.02, len(wavelength))
combined = profile1 + profile2 + noise
# If you want to plot what this looks like
pl.plot(wavelength, combined, label="Measured")
pl.plot(wavelength, profile1, color='red', linestyle='dashed', label="1")
pl.plot(wavelength, profile2, color='green', linestyle='dashed', label="2")
pl.title("Feature One and Two")
pl.legend()
请注意,我实际要拟合的函数并不是高斯函数——因此,请使用通用函数(例如我的示例中的GaussFunc),而不是“内置”的pymc.Normal()类型函数来提供示例。
此外,我了解模型选择是另一个问题:因此,在当前噪声下,可能只有1个分量(轮廓)是统计上证明的。但我想看看1、2、3等分量的最佳解决方案。
我也不固执于使用PyMC——如果scikit-learn、astroML或其他某个包似乎完美,请告诉我!
编辑:
sigma_mc_one = mc.Uniform('sig', 0.01, 6.5)
height_mc_one = mc.Uniform('height', 0.1, 2.5)
centroid_mc_one = mc.Uniform('cen', 5015., 5040.)
但我无法构建一个可行的mc.model。
mc.Uniform('sig', 0.01, 6.5),但是从安全角度考虑,最好将范围从0开始而不是0.01。数学上,0确实会破坏模型,但在MCMC算法中,0也永远不会被选中。包括0(到0.01)的原因是为了包含真实值在0到0.01之间的小概率。 - Cam.Davidson.Pilon