我有一张大小为RGB
现在,我想将其分类到另一张图像上训练的某些颜色,并计算它们各自的AB表示:
现在,为了将每个像素分类/标记到1-7个聚类中,我目前执行以下操作(伪代码):
然而,由于图像分辨率和我手动循环每个x和y,这非常慢(52秒)。
是否有一些内置函数可以执行相同的工作?肯定有。
总结一下:我需要一种分类方法,将像素图像分类到已定义的一组聚类中。
uint8(576,720,3)的图像,我想将每个像素分类到一组颜色中。我使用rgb2lab从RGB转换为LAB空间,然后删除了L层,因此现在它是一个double(576,720,2),由AB组成。现在,我想将其分类到另一张图像上训练的某些颜色,并计算它们各自的AB表示:
Cluster 1: -17.7903 -13.1170
Cluster 2: -30.1957 40.3520
Cluster 3: -4.4608 47.2543
Cluster 4: 46.3738 36.5225
Cluster 5: 43.3134 -17.6443
Cluster 6: -0.9003 1.4042
Cluster 7: 7.3884 11.5584
现在,为了将每个像素分类/标记到1-7个聚类中,我目前执行以下操作(伪代码):
clusters;
for each x
for each y
ab = im(x,y,2:3);
dist = norm(ab - clusters); // norm of dist between ab and each cluster
[~, idx] = min(dist);
end
end
然而,由于图像分辨率和我手动循环每个x和y,这非常慢(52秒)。
是否有一些内置函数可以执行相同的工作?肯定有。
总结一下:我需要一种分类方法,将像素图像分类到已定义的一组聚类中。
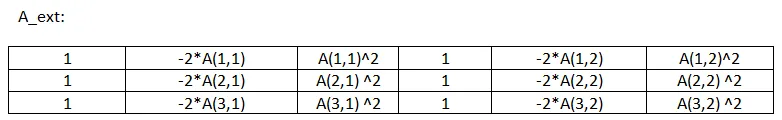
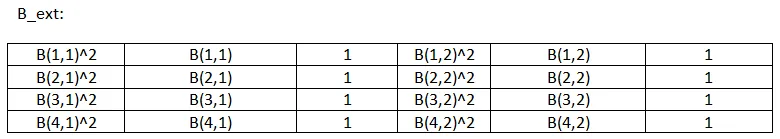

bsxfun! - Divakar