我在pandas中有一个大小为(106x106)的大型相关系数矩阵,具有以下结构:
+---+-------------------+------------------+------------------+------------------+------------------+-----------------+------------------+------------------+------------------+-------------------+
| | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 |
+---+-------------------+------------------+------------------+------------------+------------------+-----------------+------------------+------------------+------------------+-------------------+
| 0 | 1.0 | 0.465539925807 | 0.736955649673 | 0.733077703346 | -0.177380436347 | -0.268022641963 | 0.0642473239514 | -0.0136866435594 | -0.025596700815 | -0.00385065532308 |
| 1 | 0.465539925807 | 1.0 | -0.173472213691 | -0.16898620433 | -0.0460674481563 | 0.0994673318696 | 0.137137216943 | 0.061999118034 | 0.0944808695878 | 0.0229095105328 |
| 2 | 0.736955649673 | -0.173472213691 | 1.0 | 0.996627003263 | -0.172683935315 | -0.33319698831 | -0.0562591684255 | -0.0306820050477 | -0.0657065745626 | -0.0457836647012 |
| 3 | 0.733077703346 | -0.16898620433 | 0.996627003263 | 1.0 | -0.153606414649 | -0.321562257834 | -0.0465540370732 | -0.0224318843281 | -0.0586629098513 | -0.0417237678539 |
| 4 | -0.177380436347 | -0.0460674481563 | -0.172683935315 | -0.153606414649 | 1.0 | 0.0148395123941 | 0.191615549534 | 0.289211355855 | 0.28799868259 | 0.291523969899 |
| 5 | -0.268022641963 | 0.0994673318696 | -0.33319698831 | -0.321562257834 | 0.0148395123941 | 1.0 | 0.205432455075 | 0.445668299971 | 0.454982398693 | 0.427323555674 |
| 6 | 0.0642473239514 | 0.137137216943 | -0.0562591684255 | -0.0465540370732 | 0.191615549534 | 0.205432455075 | 1.0 | 0.674329392219 | 0.727261969241 | 0.67891326835 |
| 7 | -0.0136866435594 | 0.061999118034 | -0.0306820050477 | -0.0224318843281 | 0.289211355855 | 0.445668299971 | 0.674329392219 | 1.0 | 0.980543049288 | 0.939548790275 |
| 8 | -0.025596700815 | 0.0944808695878 | -0.0657065745626 | -0.0586629098513 | 0.28799868259 | 0.454982398693 | 0.727261969241 | 0.980543049288 | 1.0 | 0.930281915882 |
| 9 | -0.00385065532308 | 0.0229095105328 | -0.0457836647012 | -0.0417237678539 | 0.291523969899 | 0.427323555674 | 0.67891326835 | 0.939548790275 | 0.930281915882 | 1.0 |
+---+-------------------+------------------+------------------+------------------+------------------+-----------------+------------------+------------------+------------------+-------------------+
为了简便起见,此处截断。
如果我计算关联性并使用以下代码绘制树状图:
from scipy.cluster.hierarchy import dendrogram, linkage
Z = linkage(result_df.corr(),'average')
plt.figure()
fig, axes = plt.subplots(1, 1, figsize=(20, 20))
axes.tick_params(axis='both', which='major', labelsize=15)
dendrogram(Z=Z, labels=result_df_null_cols.columns,
leaf_rotation=90., ax=axes,
color_threshold=2.)
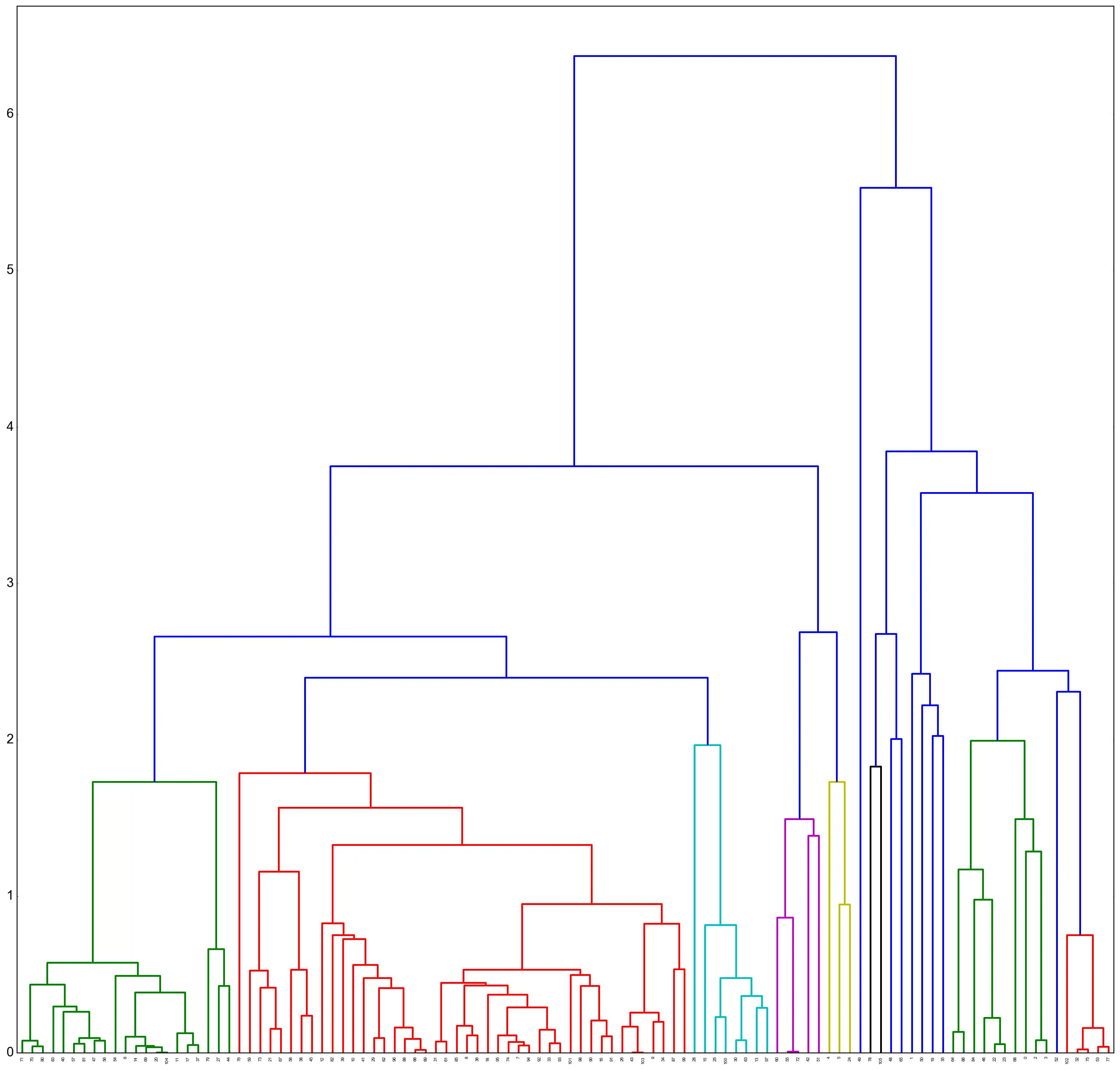 我的问题涉及y轴。在我看到的所有示例中,Y轴被限制在0和2之间 - 我已经理解它表示为
我的问题涉及y轴。在我看到的所有示例中,Y轴被限制在0和2之间 - 我已经理解它表示为(1-corr)。在我的结果中,边界要高得多。0代表高度相关的项目(1-1=0),而2则是低度相关内容的截止点(1--1=2)。我找到了以下答案,但它与这个答案不一致,以及参考讲义在这里。
无论如何 - 希望有人可以澄清哪个来源是正确的,并帮助传播一些关于此主题的知识。