前言
本周我遇到了这个问题,唯一与之相关的问题是这篇帖子。我的需求几乎与OP相同:
但我还需要:
- 能够执行统计检验;
- 同时符合
scipy随机变量接口的规范。
正如@Jacques Gaudin所指出的,rv_continous的接口(详见分布结构)并没有保证从该类继承时要遵循loc和scale参数,而这在某种程度上是误导性的和不幸的。
当然,通过实现__init__方法可以创建缺失的绑定,但这是一个折衷方案:它打破了scipy目前用于实现随机变量的模式(其中一个实现的例子为对数正态分布)。
因此,我花时间深入研究了scipy代码,并为这个分布创建了一个MCVE。虽然它不是完全完整的(主要缺失了矩重写),但它满足OP和我的需求,同时具有令人满意的精度和性能。
MCVE
这个随机变量的接口遵循规范的实现可以是:
class logitnorm_gen(stats.rv_continuous):
def _argcheck(self, m, s):
return (s > 0.) & (m > -np.inf)
def _pdf(self, x, m, s):
return stats.norm(loc=m, scale=s).pdf(special.logit(x))/(x*(1-x))
def _cdf(self, x, m, s):
return stats.norm(loc=m, scale=s).cdf(special.logit(x))
def _rvs(self, m, s, size=None, random_state=None):
return special.expit(m + s*random_state.standard_normal(size))
def fit(self, data, **kwargs):
return stats.norm.fit(special.logit(data), **kwargs)
logitnorm = logitnorm_gen(a=0.0, b=1.0, name="logitnorm")
这个实现可以释放大部分
scipy随机变量的潜力。
N = 1000
law = logitnorm(0.24, 1.31)
sample = law.rvs(size=N)
params = logitnorm.fit(sample)
check = stats.kstest(sample, law.cdf)
bins = np.arange(0.0, 1.1, 0.1)
expected = np.diff(law.cdf(bins))
由于它依赖于scipy正态分布,因此我们可以假设其底层函数具有与正态随机变量对象相同的准确性和性能。但是,当处理高度倾斜的分布时,特别是在支持边界处,它可能确实会受到浮点算术不精确的影响。
测试
为了检查它的表现如何,我们绘制一些感兴趣的分布并进行检查。
让我们创建一些固定装置:
def generate_fixtures(
locs=[-2.0, -1.0, 0.0, 0.5, 1.0, 2.0],
scales=[0.32, 0.56, 1.00, 1.78, 3.16],
sizes=[100, 1000, 10000],
seeds=[789, 123456, 999999]
):
for (loc, scale, size, seed) in itertools.product(locs, scales, sizes, seeds):
yield {"parameters": {"loc": loc, "scale": scale}, "size": size, "random_state": seed}
并且对相关的分布和样本进行检查:
eps = 1e-8
x = np.linspace(0. + eps, 1. - eps, 10000)
for fixture in generate_fixtures():
parameters = fixture.pop("parameters")
normal = stats.norm(**parameters)
sample = special.expit(normal.rvs(**fixture))
law = logitnorm(m=parameters["loc"], s=parameters["scale"])
check = law.rvs(**fixture)
p = logitnorm.fit(sample)
trial = logitnorm(*p)
resample = trial.rvs(**fixture)
ks = stats.kstest(check, trial.cdf)
bins = np.histogram(resample)[1]
obs = np.diff(trial.cdf(bins))*fixture["size"]
ref = np.diff(law.cdf(bins))*fixture["size"]
chi2 = stats.chisquare(obs, ref, ddof=2)
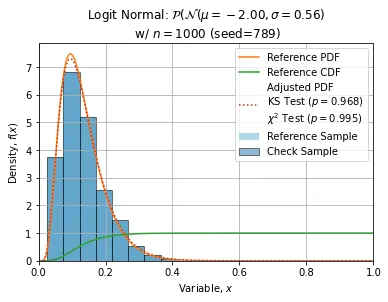
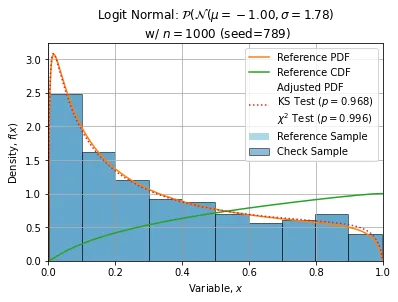
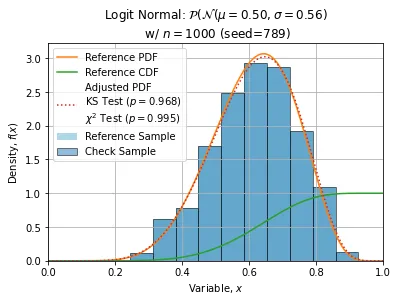
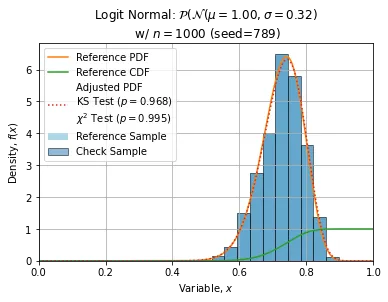
以下展示了使用n=1000, seed=789(此样本相当普通)进行的一些调整:




LogitNormal().pdf?我猜你应该调用LogitNormal()._pdf。因为_pdf是你在LogitNormal类中唯一方法的调用方式。 - user10325516_pdf或_cdf是如何对rv_continuous进行子类化的。 - mapfrv_continuous.pdf,该方法使用loc和scale参数对x值进行转换。我猜你可以使用默认值得到相同的结果:sigma, mu = 1, 0。 - user10325516rv_continuous.pdf计算概率密度函数。而scipy.stats.norm.pdf也计算概率密度函数。我想这就是我能告诉你的全部了,因为我不熟悉这些函数。祝你好运。 - user10325516