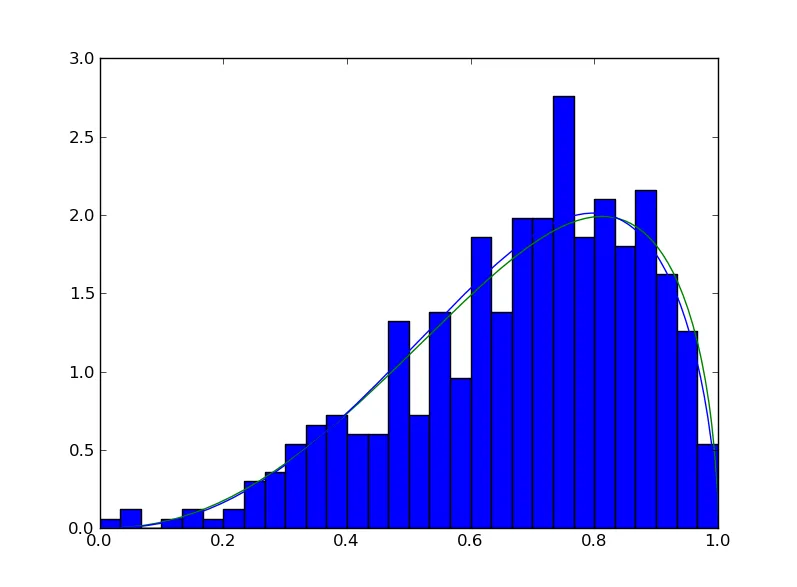
我正在尝试找到正确的拟合beta分布的方法。这不是一个真实的问题,我只是在测试几种不同方法的效果,在做这件事时有些困惑。
这是我正在处理的Python代码,我测试了三种不同的途径: 1>:使用矩(样本均值和方差)进行拟合。 2>:通过最小化负对数似然(使用scipy.optimize.fmin())进行拟合。 3>:简单地调用scipy.stats.beta.fit()。
from scipy.optimize import fmin
from scipy.stats import beta
from scipy.special import gamma as gammaf
import matplotlib.pyplot as plt
import numpy
def betaNLL(param,*args):
'''Negative log likelihood function for beta
<param>: list for parameters to be fitted.
<args>: 1-element array containing the sample data.
Return <nll>: negative log-likelihood to be minimized.
'''
a,b=param
data=args[0]
pdf=beta.pdf(data,a,b,loc=0,scale=1)
lg=numpy.log(pdf)
#-----Replace -inf with 0s------
lg=numpy.where(lg==-numpy.inf,0,lg)
nll=-1*numpy.sum(lg)
return nll
#-------------------Sample data-------------------
data=beta.rvs(5,2,loc=0,scale=1,size=500)
#----------------Normalize to [0,1]----------------
#data=(data-numpy.min(data))/(numpy.max(data)-numpy.min(data))
#----------------Fit using moments----------------
mean=numpy.mean(data)
var=numpy.var(data,ddof=1)
alpha1=mean**2*(1-mean)/var-mean
beta1=alpha1*(1-mean)/mean
#------------------Fit using mle------------------
result=fmin(betaNLL,[1,1],args=(data,))
alpha2,beta2=result
#----------------Fit using beta.fit----------------
alpha3,beta3,xx,yy=beta.fit(data)
print '\n# alpha,beta from moments:',alpha1,beta1
print '# alpha,beta from mle:',alpha2,beta2
print '# alpha,beta from beta.fit:',alpha3,beta3
#-----------------------Plot-----------------------
plt.hist(data,bins=30,normed=True)
fitted=lambda x,a,b:gammaf(a+b)/gammaf(a)/gammaf(b)*x**(a-1)*(1-x)**(b-1) #pdf of beta
xx=numpy.linspace(0,max(data),len(data))
plt.plot(xx,fitted(xx,alpha1,beta1),'g')
plt.plot(xx,fitted(xx,alpha2,beta2),'b')
plt.plot(xx,fitted(xx,alpha3,beta3),'r')
plt.show()
我面临的问题是关于归一化过程(
z=(x-a)/(b-a)),其中a和b分别是样本的最小值和最大值。
当我不进行归一化时,所有的东西都正常工作,不同的拟合方法之间有轻微的差异,但是还是相当好的。
但是当我进行了归一化后,这就是我得到的结果图。
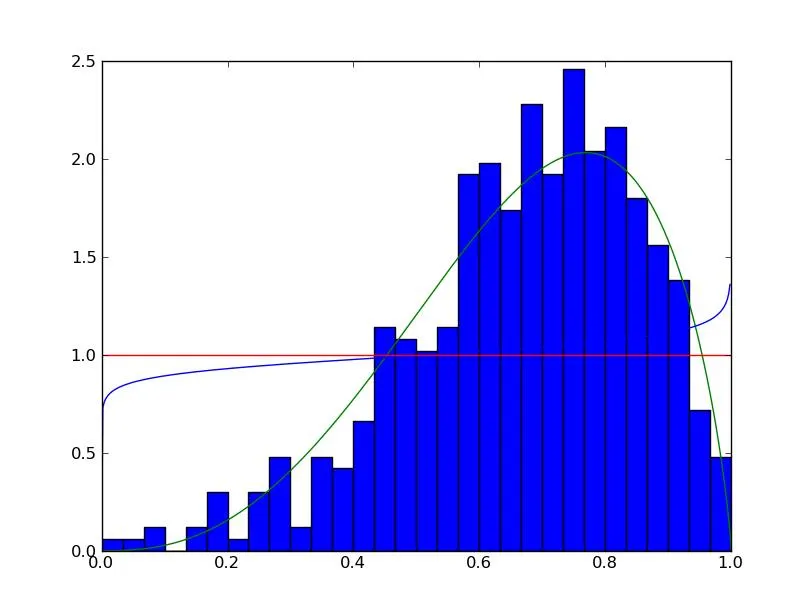
只有矩法(绿线)看起来还可以。
scipy.stats.beta.fit()方法(红线)始终是均匀的,无论我用什么参数生成随机数。
而MLE(蓝线)失败了。
因此,似乎归一化正在引起这些问题。但我认为在beta分布中,具有x=0和x=1是合法的。如果给定一个真实的世界问题,那么将样本观测值归一化使其在[0,1]之间不是第一步吗?在这种情况下,我应该如何拟合曲线?