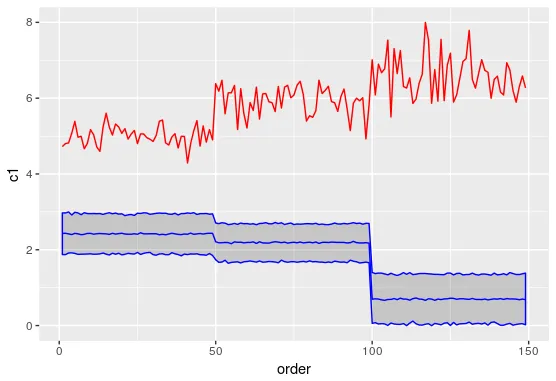
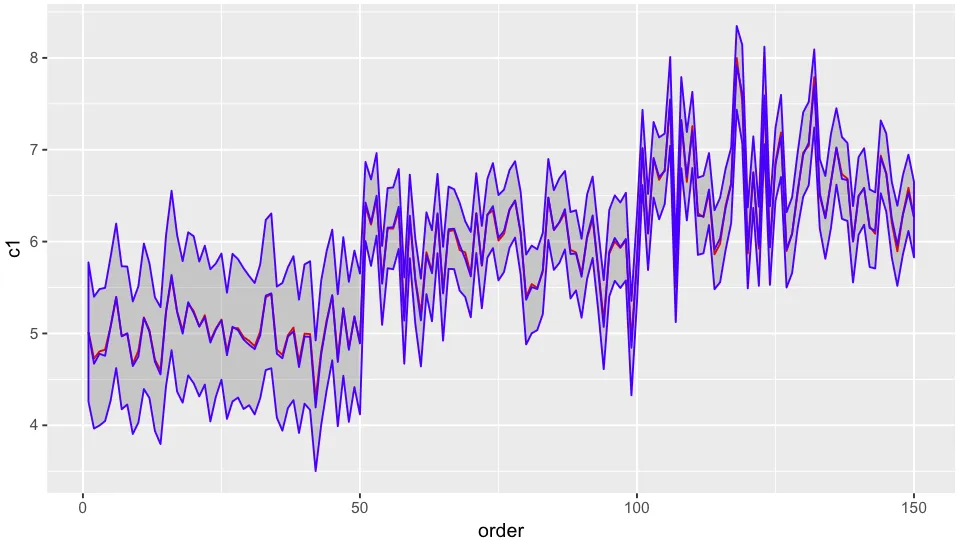
我正在尝试获取混合模型预测的置信区间。predict函数不会输出任何置信区间。一些StackOverflow答案建议使用merTools包中的predictInterval函数来获取区间,但是这两个函数的预测估计存在差异,我正在尝试在下面的图中进行比较。请问有人能够友好地告诉我这里出了什么问题吗?另外,我正在尝试构建的实际模型与下面代码段中所示的模型相似,其中除截距外没有固定效应分量。
红线表示预测的 'c1',蓝线表示预测的 'c2'。
library(merTools)
library(lme4)
dat <- iris
mod <- lmer(Sepal.Length ~ 1 + (1 + Sepal.Width + Petal.Length +
Petal.Width|Species), data=dat)
c1 <- predict(mod, dat)
c2 <- predictInterval(mod, dat)
plot_data <- cbind(c1, c2)
plot_data$order <- c(1:nrow(plot_data))
library(ggplot2)
ggplot(plot_data) + geom_line(aes(x=order, y=c1), color='red') +
geom_ribbon(aes(x=order, ymin=lwr, ymax=upr), color='blue', alpha=0.2) +
geom_line(aes(x=order, y=fit), color='blue')
红线表示预测的 'c1',蓝线表示预测的 'c2'。