我使用了
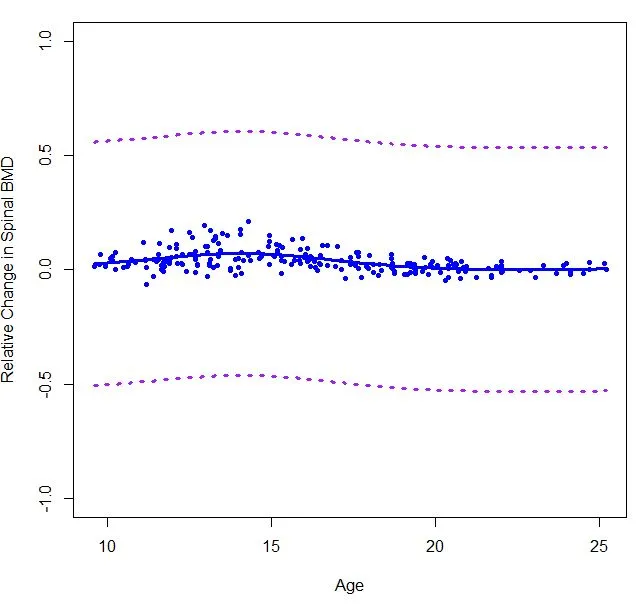
smooth.spline来估算我的数据的三次样条。但是当我使用方程计算90%点间置信区间时,结果似乎有点不对。请问是否有人能告诉我我做错了什么?我想知道是否有一个函数可以自动计算与smooth.spline函数相关的点间置信带。boneMaleSmooth = smooth.spline( bone[males,"age"], bone[males,"spnbmd"], cv=FALSE)
error90_male = qnorm(.95)*sd(boneMaleSmooth$x)/sqrt(length(boneMaleSmooth$x))
plot(boneMaleSmooth, ylim=c(-0.5,0.5), col="blue", lwd=3, type="l", xlab="Age",
ylab="Relative Change in Spinal BMD")
points(bone[males,c(2,4)], col="blue", pch=20)
lines(boneMaleSmooth$x,boneMaleSmooth$y+error90_male, col="purple",lty=3,lwd=3)
lines(boneMaleSmooth$x,boneMaleSmooth$y-error90_male, col="purple",lty=3,lwd=3)
mgcv包中的gam()函数。它立即给出了置信区间,但我不确定它是90%还是95%的置信区间或其他什么。如果有人能解释一下就太好了。males=gam(bone[males,c(2,4)]$spnbmd ~s(bone[males,c(2,4)]$age), method = "GCV.Cp")
plot(males,xlab="Age",ylab="Relative Change in Spinal BMD")
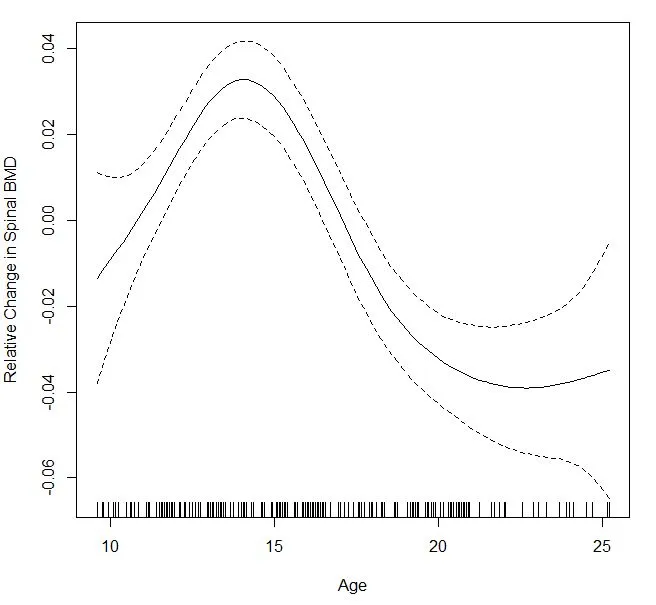
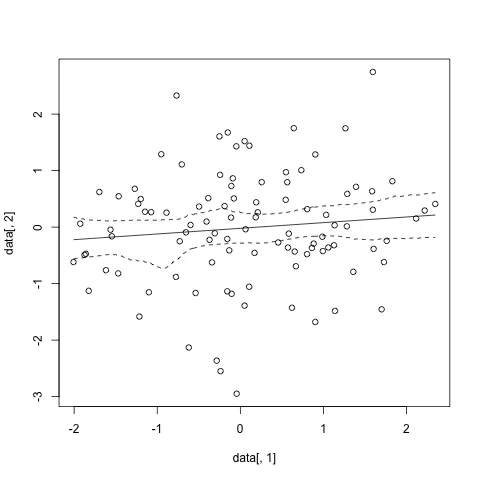
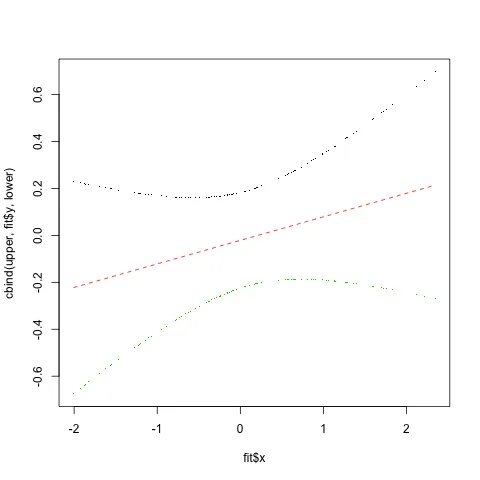
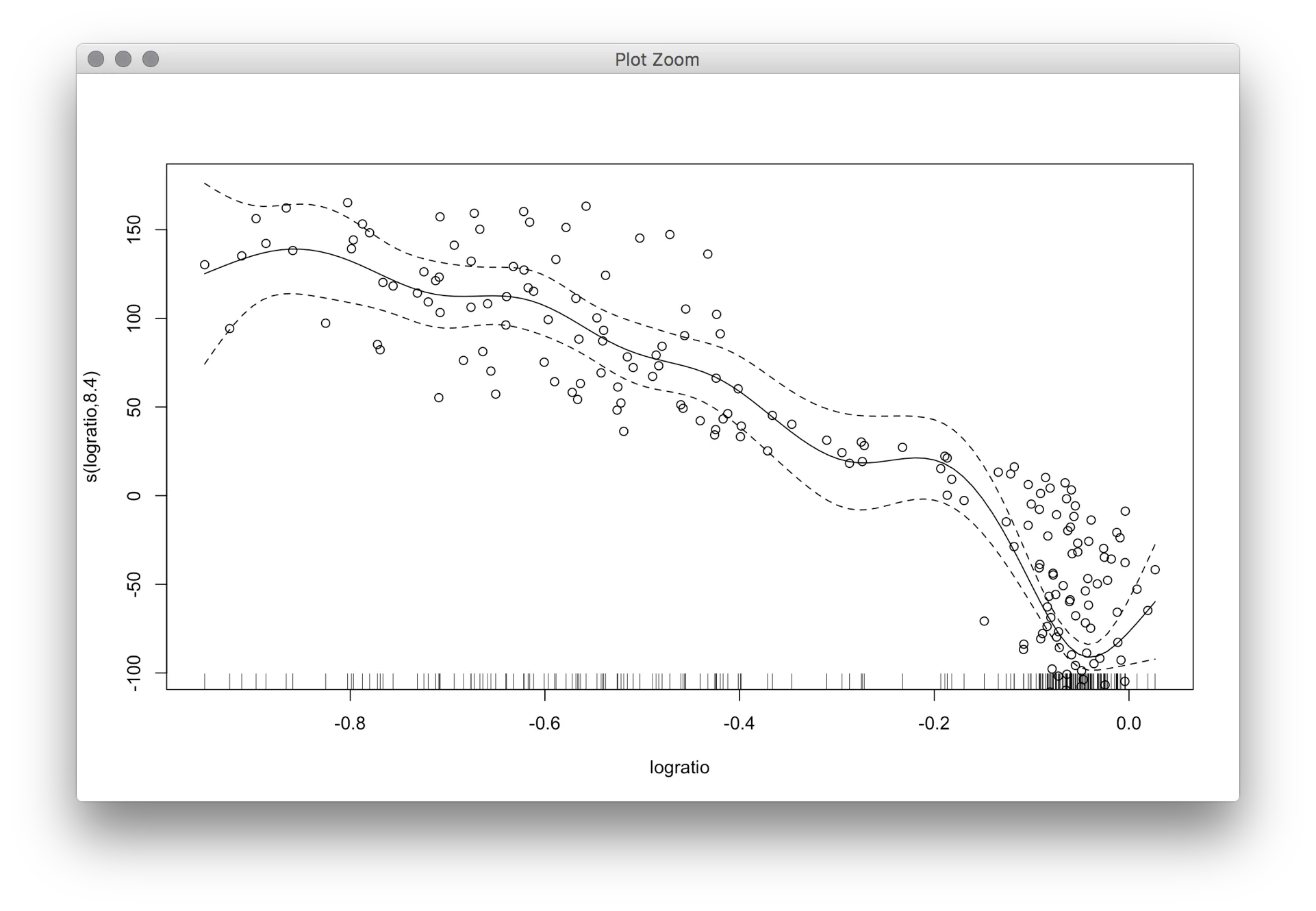
gam()中得到的结果完全不同。使用jackknife剩余项的方法也给出了非常独特的图案,并且这个绘图中的CIs非常崎岖。 - Yu Dengfit $ lev来计算区间宽度背后的逻辑吗? - foreignvol