igraph中,应用模块化算法找到图形社区后,我想绘制一个网络布局,清晰地显示不同的社区及其连接。类似于Cytoscape中的“组属性布局”:我想将每个组/社区的成员彼此靠近,并在组/社区之间保持一定的距离。我没有在igraph中找到任何提供此功能的函数。在发布这个问题时,我已经找到了一个简单的DIY解决方案,我将把它作为答案发布。但是我想知道是否有更好的可能性或更详细的解决方案?weight.community=function(row,membership,weigth.within,weight.between){
if(as.numeric(membership[which(names(membership)==row[1])])==as.numeric(membership[which(names(membership)==row[2])])){
weight=weigth.within
}else{
weight=weight.between
}
return(weight)
}
只需将其应用于您的图的边缘矩阵行(由 get.edgelist(your_graph) 给出),即可设置新的边缘权重(成员资格是来自任何社区检测算法结果的成员资格向量):
E(g)$weight=apply(get.edgelist(g),1,weight.community,membership,10,1)
然后,只需使用接受边权重的布局算法,例如Gabor建议的fruchterman.reingold。您可以调整权重参数以获得所需的图形。例如:
E(g)$weight=apply(get.edgelist(g),1,weight.community,membership,10,1)
g$layout=layout.fruchterman.reingold(g,weights=E(g)$weight)
plot(g)
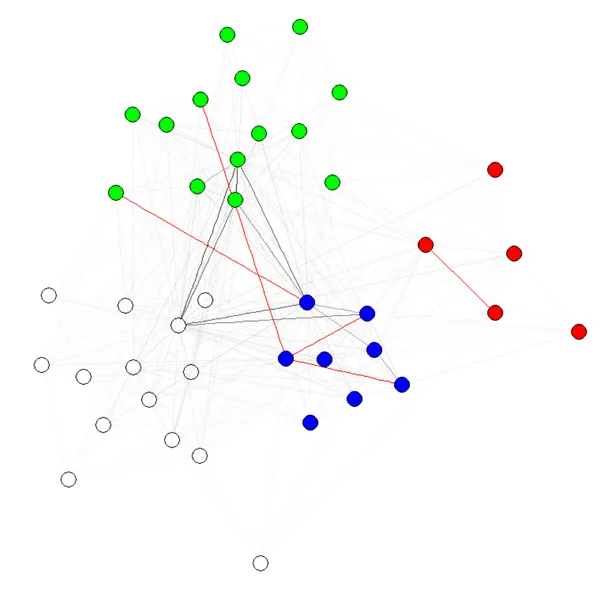
E(g)$weight=apply(get.edgelist(g),1,weight.community,membership,1000,1)
g$layout=layout.fruchterman.reingold(g,weights=E(g)$weight)
plot(g)
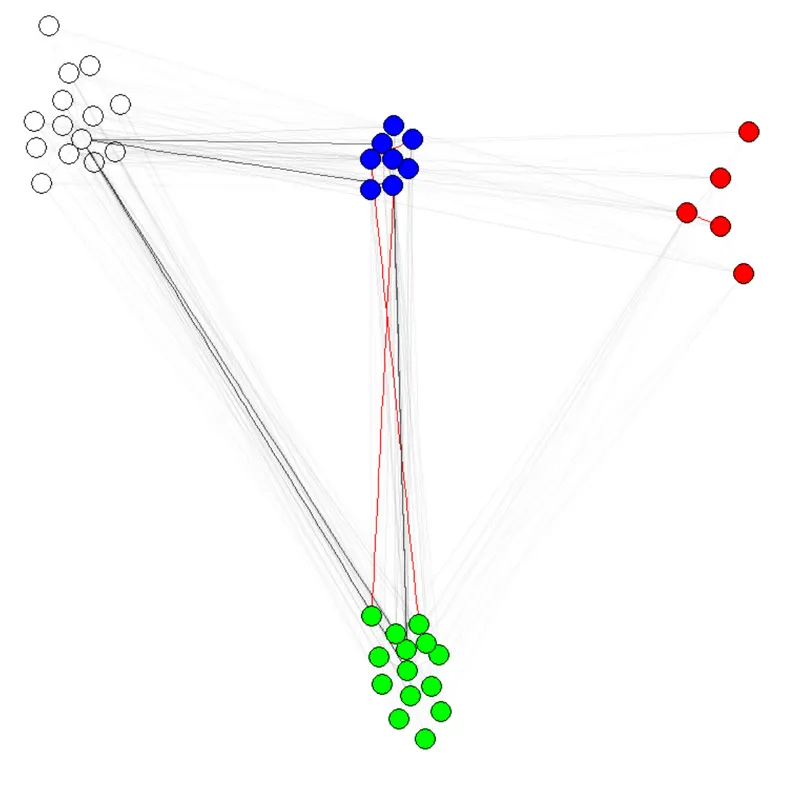
注意1:边的透明度/颜色是我的图表的其他参数。我将节点按社区着色以显示它确实起作用。
注意2:请务必使用membership(comm)而不是comm $ membership,其中comm是社区检测算法的结果(例如comm = leading.eigenvector.community(g) )。原因是在第一种情况下,您会得到一个带有名称的数字向量(我们想要的),而在第二种情况下,您会得到相同的向量但没有名称。
要获取多个社区检测算法的共识,请参见此函数。
受安托万的建议启发,我创建了这个函数:
edge.weights <- function(community, network, weight.within = 100, weight.between = 1) {
bridges <- crossing(communities = community, graph = network)
weights <- ifelse(test = bridges, yes = weight.between, no = weight.within)
return(weights)
}
这个函数的作用是:将你的社区对象放入社区槽中,将你的图形放入网络槽中。我会保留weight.between = 1,并调整weight.within的值。
然后将权重转移到节点的weight槽中:
E(graph)$weight <- edge.weights(community, graph)
最后使用一种类似于 layout_with_fr(在 igraph 1.0.1 中更名为 fruchterman.reingold)的权重布局算法。
我以 Zachary 的空手道俱乐部网络为例。
library(igraph)
library(igraphdata)
#I load the network
data(karate)
#for reproducible purposes
set.seed(23548723)
karateLayout <- layout_with_fr(karate)
par(mar = c(0,0,2,0))
plot(karate, vertex.size = 10, vertex.color = "steelblue4", edge.width = 1,
vertex.label = NA, edge.color = "darkgrey", layout = karateLayout,
main = "Zachary's karate club network" )
我使用cluster_louvain函数进行模块度的多级优化,以检测社区:
Communitykarate <- cluster_louvain(karate)
prettyColors <- c("turquoise4", "azure4", "olivedrab","deeppink4")
communityColors <- prettyColors[membership(Communitykarate)]
plot(x = Communitykarate, y = karate, edge.width = 1, vertex.size = 10,
vertex.label = NA, mark.groups = NULL, layout = karateLayout, col = communityColors,
main = "Communities in Zachary's karate club network",
edge.color = c("darkgrey","tomato2")crossing(Communitykarate, karate) + 1])
现在,这个问题存在的原因是什么。
E(karate)$weight <- edge.weights(Communitykarate, karate)
# I use the original layout as a base for the new one
karateLayoutA <- layout_with_fr(karate, karateLayout)
# the graph with the nodes grouped
plot(x = Communitykarate, y = karate, edge.width = 1, vertex.size = 10,
mark.groups = NULL, layout = karateLayoutA, vertex.label = NA, col = communityColors,
c("darkgrey","tomato2")[crossing(Communitykarate, karate) + 1],
main = "Communities in Zachary's karate club network (grouped)")
如果您尝试使用更多的重量,您将会有:
E(karate)$weight <- edge.weights(Communitykarate, karate, weight.within = 1000)
karateLayoutB <- layout_with_fr(karate, karateLayout)
plot(x = Communitykarate, y = karate, edge.width = 1, vertex.size = 10,
mark.groups = NULL, layout = karateLayoutB, vertex.label = NA, col = communityColors,
c("darkgrey","tomato2")[crossing(Communitykarate, karate) + 1],
main = "Communities in Zachary's karate club network (grouped)")
layout.modular函数从任何igraph社区检测方法的结果中提供一种分组布局的图形:
c <- fastgreedy.community(G)
layout.modular <- function(G,c){
nm <- length(levels(as.factor(c$membership)))
gr <- 2
while(gr^2<nm){
gr <- gr+1
}
i <- j <- 0
for(cc in levels(as.factor(c$membership))){
F <- delete.vertices(G,c$membership!=cc)
F$layout <- layout.kamada.kawai(F)
F$layout <- layout.norm(F$layout, i,i+0.5,j,j+0.5)
G$layout[c$membership==cc,] <- F$layout
if(i==gr){
i <- 0
if(j==gr){
j <- 0
}else{
j <- j+1
}
}else{
i <- i+1
}
}
return(G$layout)
}
G$layout <- layout.modular(G,c)
V(G)$color <- rainbow(length(levels(as.factor(c$membership))))[c$membership]
plot(G)
一种解决方案是根据模块化设置图的边权重。将模块内部的边设置为较大的权重,将模块间的边设置为较小的权重。然后调用 layout.fruchterman.reingold() 或任何支持边权重的算法。
您可能需要对实际权重值进行一些调整,因为这取决于您的图形。
library(igraph); G <- barabasi.game(100, directed = FALSE)。错误信息显示:Error in G$layout[c$membership == cc, ] <- F$layout : incorrect number of subscripts on matrix。 - majomG$layout为空。如果您使用正确维度的矩阵填充它,或者只需使用G$layout <- layout.fruchterman.reingold(G),那么代码就可以正常运行了。 - deeenes