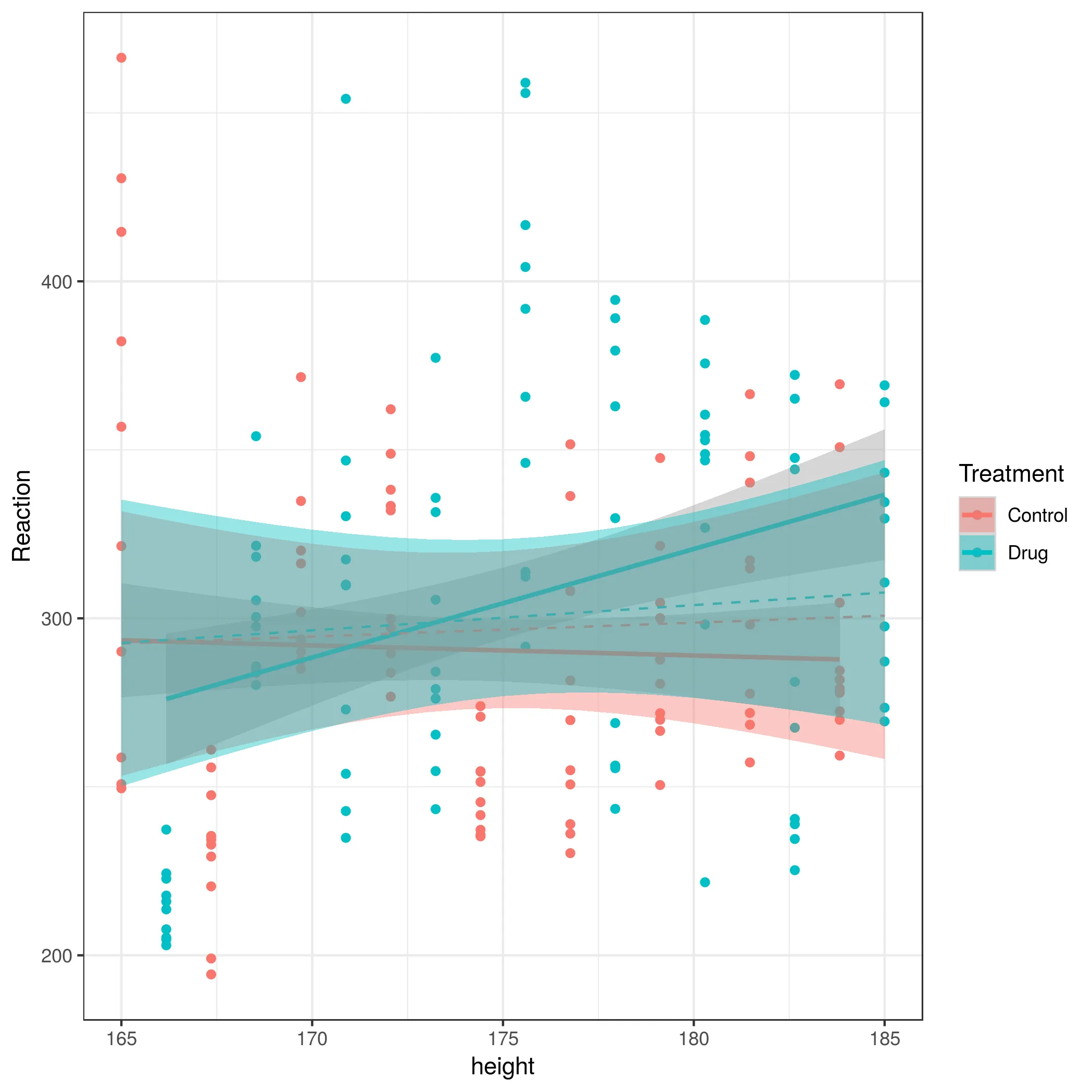
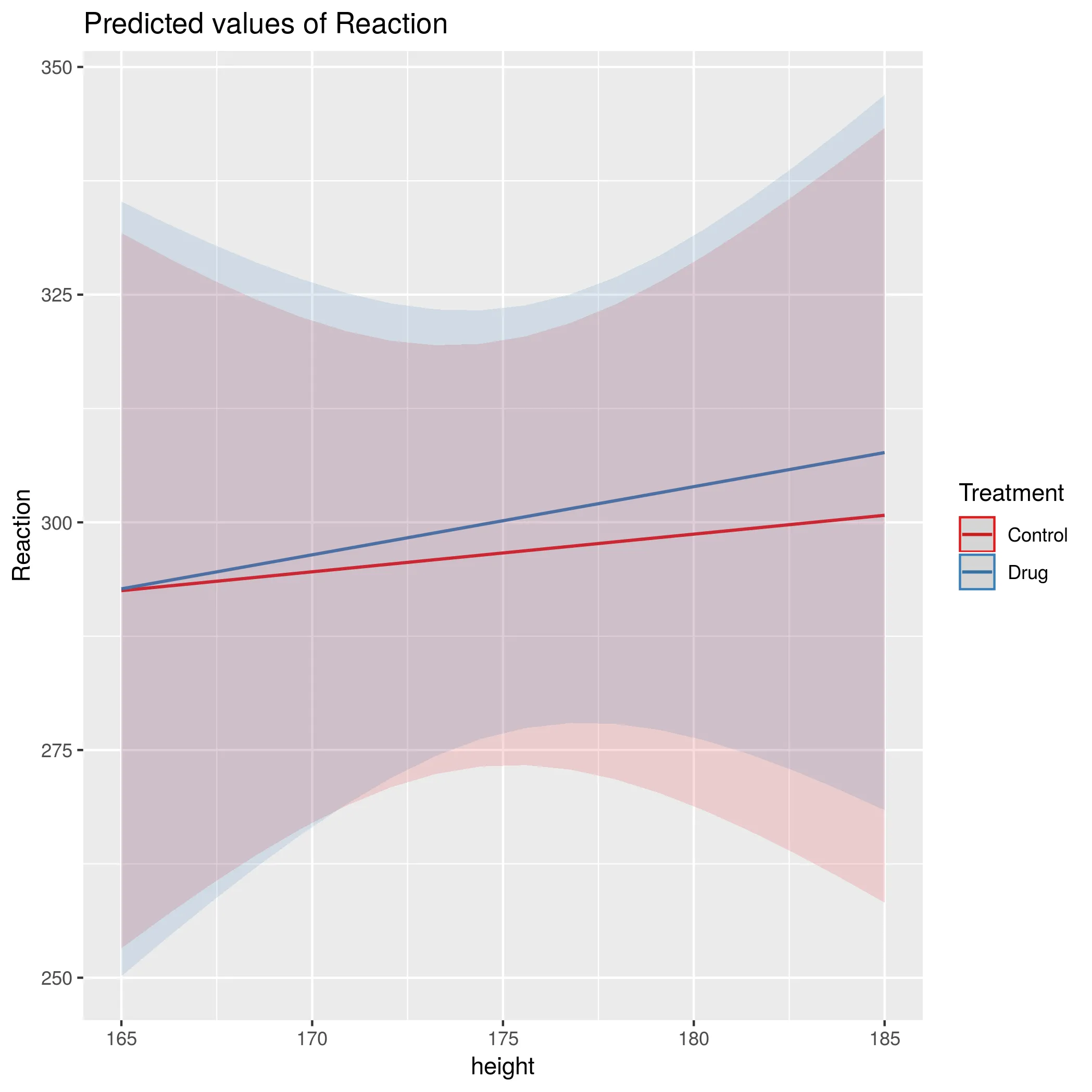
我想知道为什么在从线性混合效应模型计算95%置信带时,ggplot2生成的带比手动计算的带更窄,例如按照Ben Bolker在这里的方法进行计算predictions的置信区间。也就是说,ggplot2是否给出了模型的不准确表示?
下面是使用sleepstudy数据集的可重现示例(已修改为与我正在处理的df结构相似):
生成模型,将预测结果添加到原始数据框中,并绘制图表。
下面是使用sleepstudy数据集的可重现示例(已修改为与我正在处理的df结构相似):
data("sleepstudy") # load dataset
height <- seq(165, 185, length.out = 18) # create vector called height
Treatment <- rep(c("Control", "Drug"), 9) # create vector called treatment
Subject <- levels(sleepstudy$Subject) # get vector of Subject
ht.subject <- data.frame(height, Subject, Treatment)
sleepstudy <- dplyr::left_join(sleepstudy, ht.subject, by="Subject") # Append df so that each subject has its own height and treatment
sleepstudy$Treatment <- as.factor(sleepstudy$Treatment)
生成模型,将预测结果添加到原始数据框中,并绘制图表。
m.sleep <- lmer(Reaction ~ Treatment*height + (1 + Days|Subject), data=sleepstudy)
sleepstudy$pred <- predict(m.sleep)
ggplot(sleepstudy, aes(height, pred, col=Treatment)) + geom_smooth(method="lm")[2]
使用Bolker方法计算置信区间
newdf <- expand.grid(height=seq(165, 185, 1),
Treatment=c("Control","Drug"))
newdf$Reaction <- predict(m.sleep, newdf, re.form=NA)
modmat <- model.matrix(terms(m.sleep), newdf)
pvar1 <- diag(modmat %*% tcrossprod(vcov(m.sleep), modmat))
tvar1 <- pvar1+VarCorr(m.sleep)$Subject[1]
cmult <- 1.96
newdf <- data.frame(newdf
,plo = newdf$Reaction-cmult*sqrt(pvar1)
,phi = newdf$Reaction+cmult*sqrt(pvar1)
,tlo = newdf$Reaction-cmult*sqrt(tvar1)
,thi = newdf$Reaction+cmult*sqrt(tvar1))
# plot confidence intervals
ggplot(newdf, aes(x=height, y=Reaction, colour=Treatment)) +
geom_point() +
geom_ribbon(aes(ymin=plo, ymax=phi, fill=Treatment), alpha=0.4)[2]