在 sklearn 或其他 Python 包中是否可以获得针对 周期性 数据的 KDE?
以下是一个简化示例:我从两个正态分布中创建了一个模拟数据集,并将其映射到从0到20的区间。
import numpy as np
import matplotlib.pyplot as plt
# create dataset
data = np.hstack((np.random.normal(8, 2, 200), np.random.normal(19, 4, 200))) % 20
当我绘制KDE的结果时
# fit
from sklearn.neighbors import KernelDensity
kde = KernelDensity(bandwidth=1, kernel='gaussian')
kde.fit(data[:, None])
# plot
x_d = np.linspace(0, 20, 100)
logprob = kde.score_samples(x_d[:, None])
plt.fill_between(x_d, np.exp(logprob), alpha=0.5)
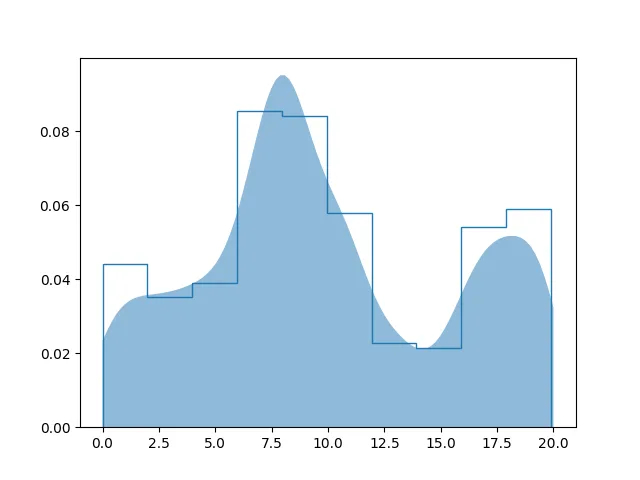
plt.hist(data, histtype = "step", density = True)
plt.ion()
plt.show()
显然,IT不知道数据的周期性:
从估计结果可以看出,有三个峰值并且在边界处不光滑。