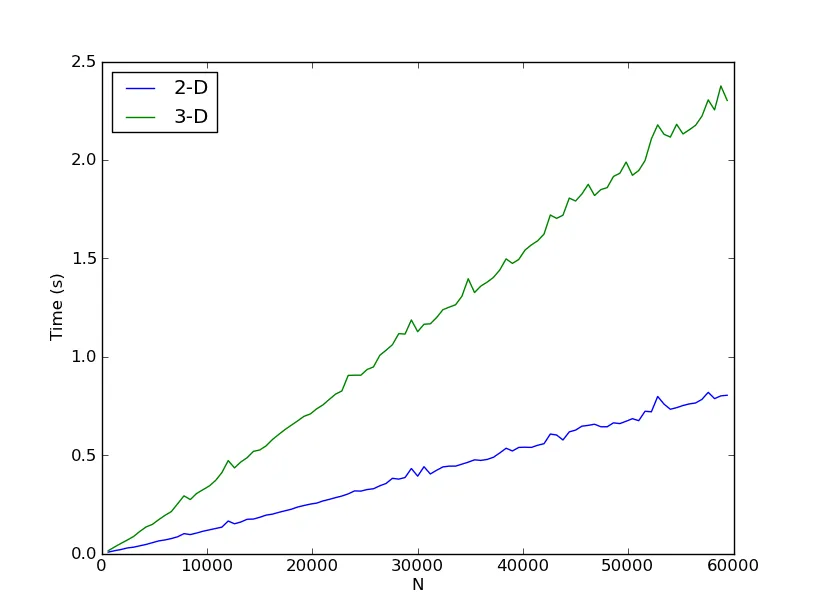
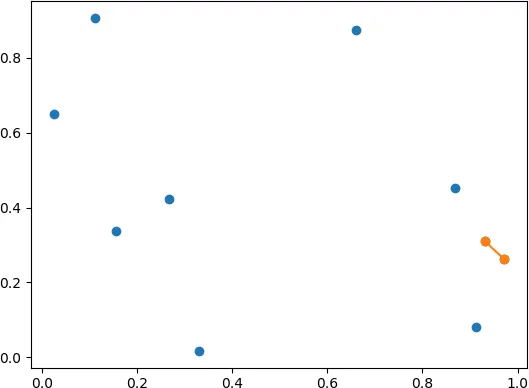
我有一组n维点,想找出其中距离最近的两个点。对于二维情况,我能想到的最好方法是:
from numpy import *
myArr = array( [[1, 2],
[3, 4],
[5, 6],
[7, 8]] )
n = myArr.shape[0]
cross = [[sum( ( myArr[i] - myArr[j] ) ** 2 ), i, j]
for i in xrange( n )
for j in xrange( n )
if i != j
]
print min( cross )
这提供了
[8, 0, 1]
但是对于大型数组来说,这种方法速度太慢了。我可以应用什么样的优化方案呢?
相关问题: