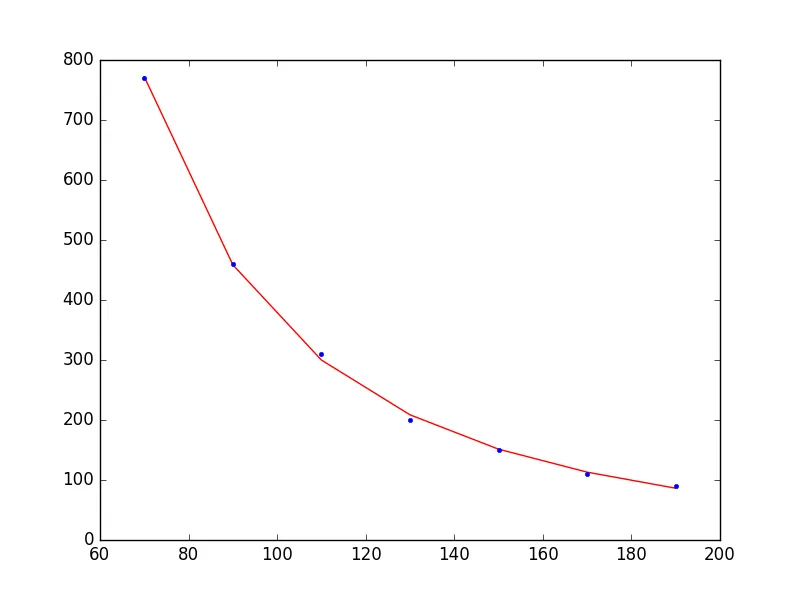
以下是一个快速但错误的答案: 您可以将您的a和b参数的误差近似为其对角线的平方根: np.sqrt(np.diagonal(pcov)),并使用参数不确定性来绘制置信区间。
该答案是错误的,因为在拟合数据到模型之前,您需要对平均disc_p1点上的误差进行估计。当进行平均时,您已经丢失了关于总体散布的信息,导致curve_fit相信您提供的y点是绝对且无可争议的。这可能会导致您的参数误差被低估。
要估计平均Y值的不确定性,您需要估计它们的离散度量,并在向curve_fit传递它时表示您的误差是绝对的。下面是一个示例,展示了如何对一个随机数据集执行此操作,其中每个点由从正态分布中抽取的1000个样本组成。
from scipy.optimize import curve_fit
import matplotlib.pylab as plt
import numpy as np
func = lambda x, a, b: a * (1 / (x**2)) + b
n_ypoints = 7
x_data = np.linspace(70, 190, n_ypoints)
n_nested_points = 1000
point_errors = 50
y_data = [func(x, 4e6, -100) + np.random.normal(x, point_errors,
n_nested_points) for x in x_data]
y_means = np.array(y_data).mean(axis = 1)
y_spread = np.array(y_data).std(axis = 1)
best_fit_ab, covar = curve_fit(func, x_data, y_means,
sigma = y_spread,
absolute_sigma = True)
sigma_ab = np.sqrt(np.diagonal(covar))
from uncertainties import ufloat
a = ufloat(best_fit_ab[0], sigma_ab[0])
b = ufloat(best_fit_ab[1], sigma_ab[1])
text_res = "Best fit parameters:\na = {}\nb = {}".format(a, b)
print(text_res)
flier_kwargs = dict(marker = 'o', markerfacecolor = 'silver',
markersize = 3, alpha=0.7)
line_kwargs = dict(color = 'k', linewidth = 1)
bp = plt.boxplot(y_data, positions = x_data,
capprops = line_kwargs,
boxprops = line_kwargs,
whiskerprops = line_kwargs,
medianprops = line_kwargs,
flierprops = flier_kwargs,
widths = 5,
manage_ticks = False)
hires_x = np.linspace(50, 190, 100)
plt.plot(hires_x, func(hires_x, *best_fit_ab), 'black')
bound_upper = func(hires_x, *(best_fit_ab + sigma_ab))
bound_lower = func(hires_x, *(best_fit_ab - sigma_ab))
plt.fill_between(hires_x, bound_lower, bound_upper,
color = 'black', alpha = 0.15)
plt.text(140, 800, text_res)
plt.xlim(40, 200)
plt.ylim(0, 1000)
plt.show()
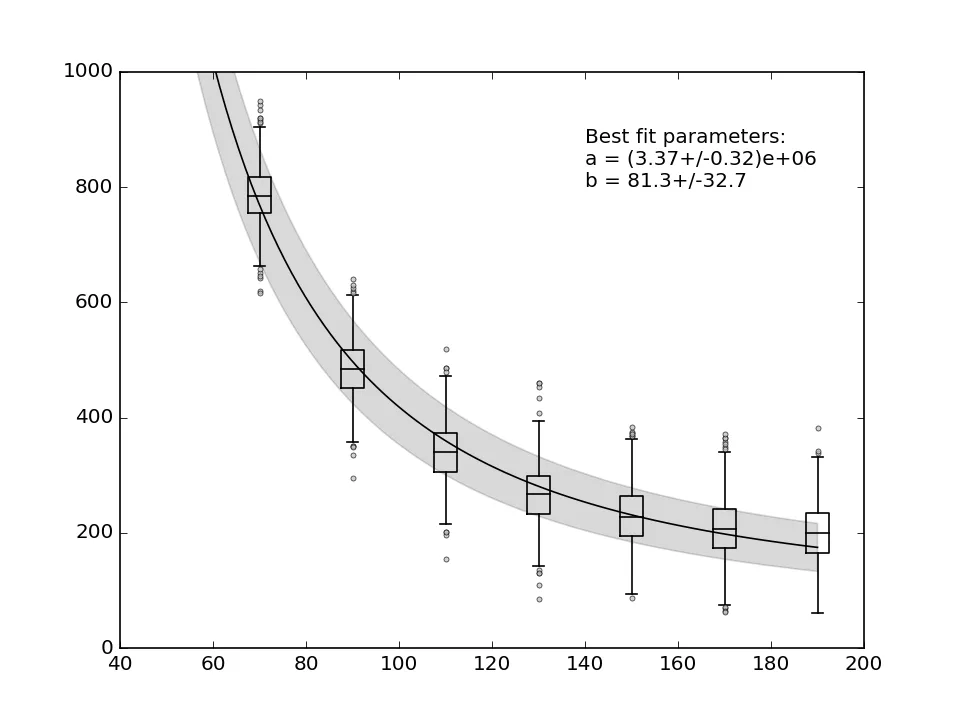
编辑:
如果您不考虑数据点上的内在误差,那么使用我之前提到的“快速而错误”的情况可能就足够了。然后可以使用协方差矩阵的对角线条目的平方根来计算置信区间。但是请注意,由于我们丢弃了不确定性,所以置信区间现在已经缩小了:
from scipy.optimize import curve_fit
import matplotlib.pylab as plt
import numpy as np
func = lambda x, a, b: a * (1 / (x**2)) + b
n_ypoints = 7
x_data = np.linspace(70, 190, n_ypoints)
y_data = np.array([786.31, 487.27, 341.78, 265.49,
224.76, 208.04, 200.22])
best_fit_ab, covar = curve_fit(func, x_data, y_data)
sigma_ab = np.sqrt(np.diagonal(covar))
from uncertainties import ufloat
a = ufloat(best_fit_ab[0], sigma_ab[0])
b = ufloat(best_fit_ab[1], sigma_ab[1])
text_res = "Best fit parameters:\na = {}\nb = {}".format(a, b)
print(text_res)
plt.scatter(x_data, y_data, facecolor = 'silver',
edgecolor = 'k', s = 10, alpha = 1)
hires_x = np.linspace(50, 200, 100)
plt.plot(hires_x, func(hires_x, *best_fit_ab), 'black')
bound_upper = func(hires_x, *(best_fit_ab + sigma_ab))
bound_lower = func(hires_x, *(best_fit_ab - sigma_ab))
plt.fill_between(hires_x, bound_lower, bound_upper,
color = 'black', alpha = 0.15)
plt.text(140, 630, text_res)
plt.xlim(60, 200)
plt.ylim(0, 800)
plt.show()
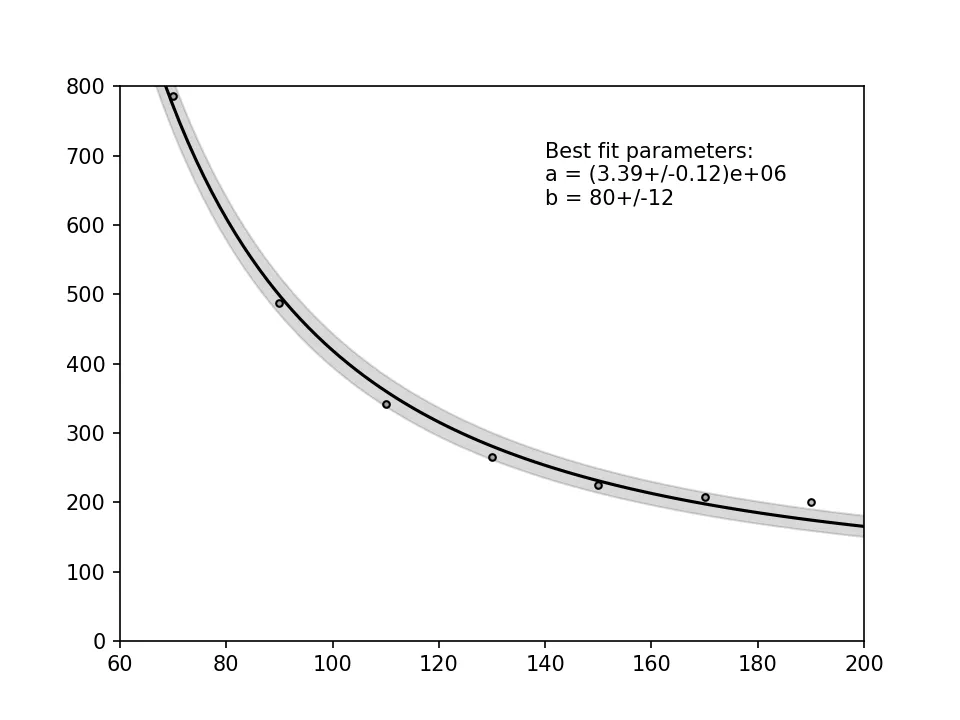
如果您不确定是否应包括绝对误差或如何在您的情况下估计它们,最好在Cross Validated上寻求建议,因为Stack Overflow主要用于回归方法实现的讨论,而不是关于底层统计学的讨论。