我有一些数据,使用scipy.stats.normal对象的fit函数进行正态分布拟合,如下所示:
import numpy as np
import matplotlib.pyplot as plt
from scipy.stats import norm
import matplotlib.mlab as mlab
x = np.random.normal(size=50000)
fig, ax = plt.subplots()
nbins = 75
mu, sigma = norm.fit(x)
n, bins, patches = ax.hist(x,nbins,normed=1,facecolor = 'grey', alpha = 0.5, label='before');
y0 = mlab.normpdf(bins, mu, sigma) # Line of best fit
ax.plot(bins,y0,'k--',linewidth = 2, label='fit before')
ax.set_title('$\mu$={}, $\sigma$={}'.format(mu, sigma))
plt.show()
我现在想提取拟合后的mu和sigma值中的不确定度/误差。我该怎么做?
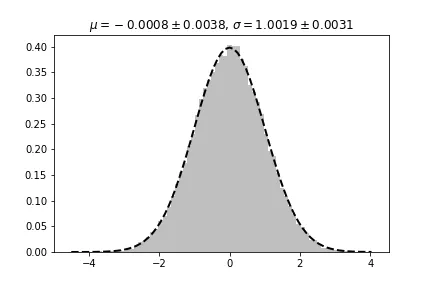
norm.fit不会受到这些不确定性的影响吗?除了报告这些不确定性之外,curve_fit和norm.fit有什么不同? - Always Learning Forevernorm.fit的作用:我不是100%确定,但我相信scipy.stats.norm.fit()使用Nelder-Mead进行拟合,而curve_fit使用Levenberg-Marquardt。我不知道scipy.stats.norm.fit()是否尝试估计不确定性,但我怀疑不会。 - M Newville