在生物学中,我们经常需要绘制剂量-反应曲线。R包“drc”非常有用,并且基本图形可以轻松处理“drm模型”。然而,我想将我的drm曲线添加到ggplot2中。
我的数据集:
我的问题是:
1. 是否有直接在ggplot2中绘制drm模型的方法? 2. 如何将数据集与其对应的4-PL曲线拟合联系起来,以便它们将以相同的颜色绘制?
我的数据集:
library("drc")
library("reshape2")
library("ggplot2")
demo=structure(list(X = c(0, 1e-08, 3e-08, 1e-07, 3e-07, 1e-06, 3e-06,
1e-05, 3e-05, 1e-04, 3e-04), Y1 = c(0, 1, 12, 19, 28, 32, 35,
39, NA, 39, NA), Y2 = c(0, 0, 10, 18, 30, 35, 41, 43, NA, 43,
NA), Y3 = c(0, 4, 15, 22, 28, 35, 38, 44, NA, 44, NA)), .Names = c("X",
"Y1", "Y2", "Y3"), class = "data.frame", row.names = c(NA, -11L
))
使用基础图形:
plot(drm(data = reshape2::melt(demo,id.vars = "X"),value~X,fct=LL.4(),na.action = na.omit),type="bars")
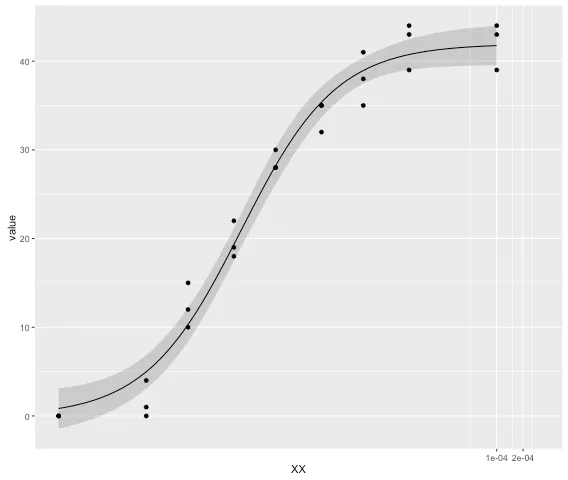
生成一个不错的四参数剂量响应曲线图。
试图在ggplot2中绘制相同的图表时,我遇到了两个问题。
There is no way of directly adding the drm model curve. I need to rewrite the 4-PL as a function and add it in the form of a stat_function, which is cumbersome to say the least.
ggplot(reshape2::melt(demo,id.vars = "X"),aes(X,value)) + geom_point() + stat_function(fun = function(x){ drm_y=function(x, drm){ coef(drm)[2]+((coef(drm)[3]-coef(drm)[2])/(1+exp((coef(drm)[1]*(log(x)-log(coef(drm)[4])))))) } + drm_y(x,drm = drm(data = reshape2::melt(demo,id.vars = "X"), value~X, fct=LL.4(), na.action = na.omit)) })If that wasn't enough it only works if scale_x is continuous. If I want to add
scale_x_log10(), I get:Warning message: In log(x): NaNs produced.
我的问题是:
1. 是否有直接在ggplot2中绘制drm模型的方法? 2. 如何将数据集与其对应的4-PL曲线拟合联系起来,以便它们将以相同的颜色绘制?