我有一个关于 geom_bar 数据顺序的问题。
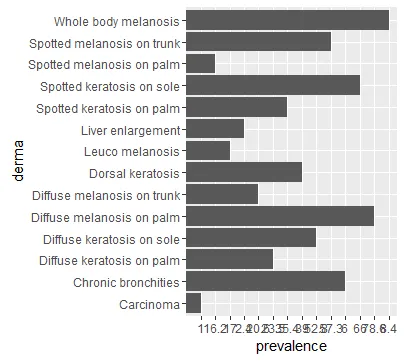
这是我的数据集:
SM_P,Spotted melanosis on palm,16.2
DM_P,Diffuse melanosis on palm,78.6
SM_T,Spotted melanosis on trunk,57.3
DM_T,Diffuse melanosis on trunk,20.6
LEU_M,Leuco melanosis,17
WB_M,Whole body melanosis,8.4
SK_P,Spotted keratosis on palm,35.4
DK_P,Diffuse keratosis on palm,23.5
SK_S,Spotted keratosis on sole,66
DK_S,Diffuse keratosis on sole,52.8
CH_BRON,Dorsal keratosis,39
LIV_EN,Chronic bronchities,6
DOR,Liver enlargement,2.4
CARCI,Carcinoma,1
我指定以下列名:
colnames(df) <- c("abbr", "derma", "prevalence") # Assign row and column names
然后我绘制:
ggplot(data=df, aes(x=derma, y=prevalence)) + geom_bar(stat="identity") + coord_flip()
为什么ggplot2会随机更改我的数据顺序。我希望数据的顺序与我的data.frame一致。
非常感谢您的任何帮助!
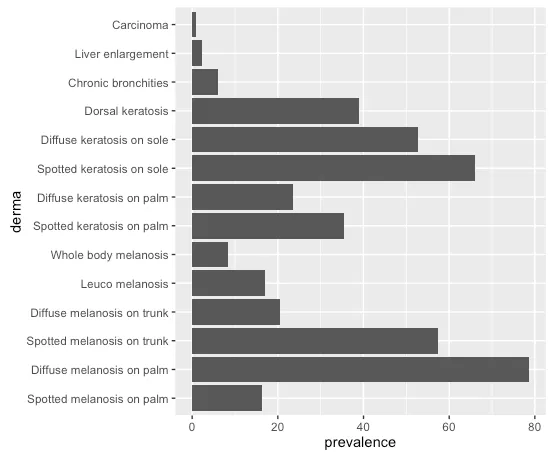
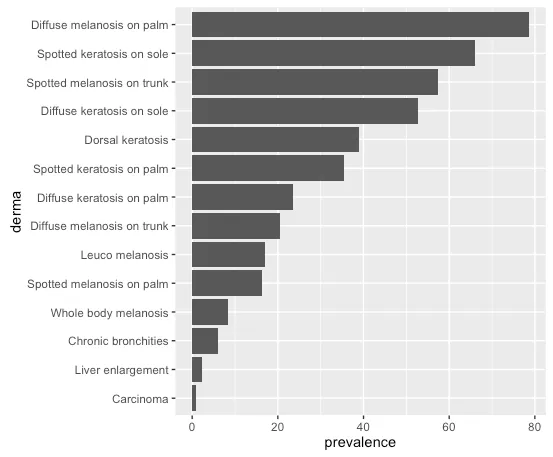
derma2因子,但随后使用了x=derma。 - arvi1000df,df $ derma和df $ derma2的顺序完全相同。所以,如果我更改绘图的df$,它并没有什么影响。 - Stückelevels(df$derma)。你可以按任意顺序放置它们以进行绘图。 - Gregor Thomas