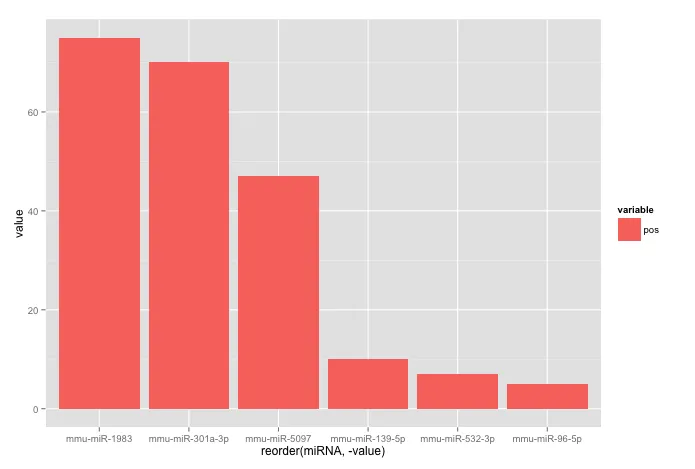
我想制作一个条形图,其中的绘图按照具有最高value的miRNA到具有最低miRNA的顺序进行排序。为什么我的代码不起作用?
> head(corr.m)
miRNA variable value
1 mmu-miR-532-3p pos 7
2 mmu-miR-1983 pos 75
3 mmu-miR-301a-3p pos 70
4 mmu-miR-96-5p pos 5
5 mmu-miR-139-5p pos 10
6 mmu-miR-5097 pos 47
ggplot(corr.m, aes(x=reorder(miRNA, value), y=value, fill=variable)) +
geom_bar(stat="identity")