我正在尝试使用scikit-learn拟合高斯函数的总和,因为与使用curve_fit相比,scikit-learn GaussianMixture似乎更加健壮。
问题:它不能很好地拟合甚至是单个高斯峰截断部分的情况:
from sklearn import mixture
import matplotlib.pyplot
import matplotlib.mlab
import numpy as np
clf = mixture.GaussianMixture(n_components=1, covariance_type='full')
data = np.random.randn(10000)
data = [[x] for x in data]
clf.fit(data)
data = [item for sublist in data for item in sublist]
rangeMin = int(np.floor(np.min(data)))
rangeMax = int(np.ceil(np.max(data)))
h = matplotlib.pyplot.hist(data, range=(rangeMin, rangeMax), normed=True);
plt.plot(np.linspace(rangeMin, rangeMax),
mlab.normpdf(np.linspace(rangeMin, rangeMax),
clf.means_, np.sqrt(clf.covariances_[0]))[0])
给出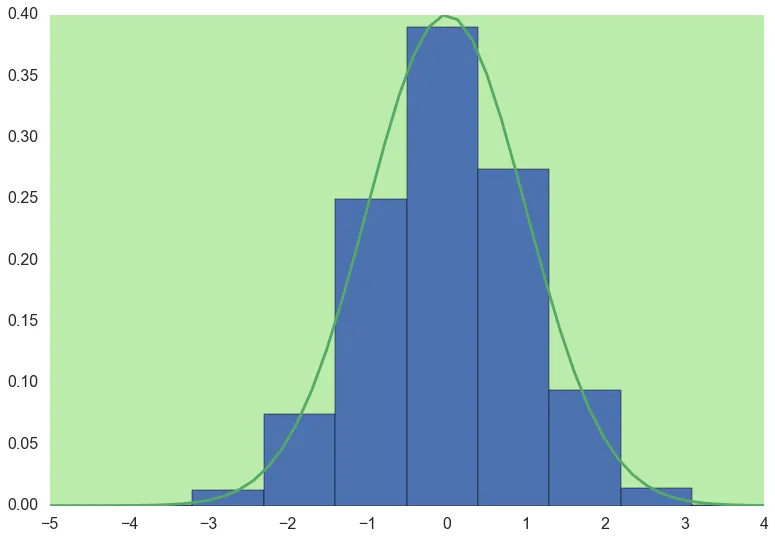 ,现在将
,现在将data = [[x] for x in data]更改为data = [[x] for x in data if x <0]以截断分布返回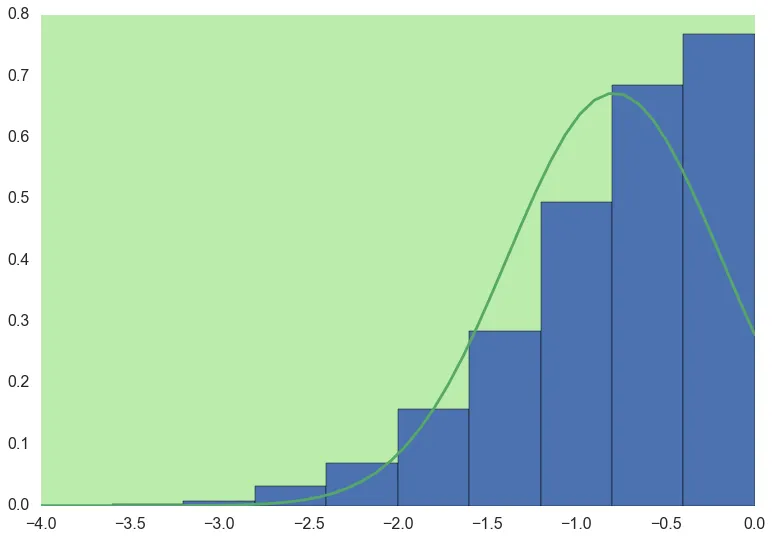 。有什么想法可以使截断适当地拟合吗?
。有什么想法可以使截断适当地拟合吗?
注意:该分布不一定在中间被截断,可能留下完整分布的50%至100%之间的任何内容。
如果有人能指向替代软件包,我也会很高兴。我只尝试过curve_fit,但在涉及两个以上峰值时无法得到有用的结果。
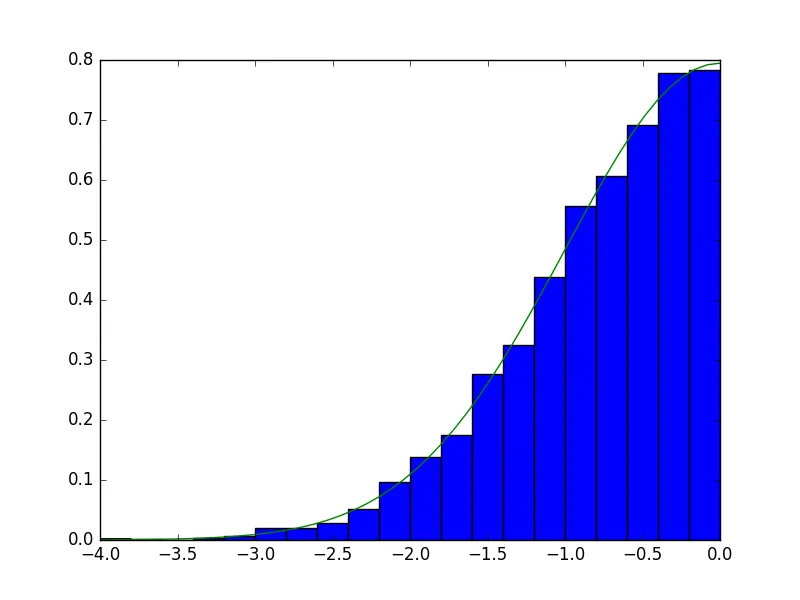
n_components=1。这就是整个想法。 - saschan_components=1... - lhcgenevascipy.optimize中的其他最小化方法吗?它是一个非常强大的工具箱,我认为不应该轻易地被忽视。 - Vlas Sokolov