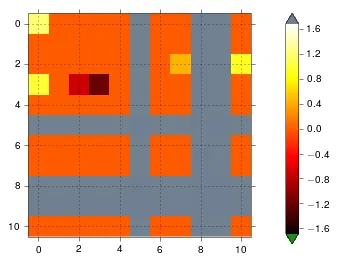
我正在生成一个热图,其中数据具有固定的异常值数量,并且我需要将这些异常值显示为颜色,超出了我使用的cmap"hot"的颜色板。使用cmap.set_bad('green')和np.ma.masked_values(data, outlier),我可以得到一个看起来正确的图形,但是即使我使用cmap.set_over('green'),颜色条也无法与数据正确同步。下面是我一直在尝试的代码:
plt.xlim(0,35)
plt.ylim(0,35)
img=plt.imshow(data, interpolation='none',norm=norm, cmap=cmap,vmax=outlier)
cb_ax=fig.add_axes([0.85, 0.1, 0.03, 0.8])
cb=mpl.colorbar.ColorbarBase(cb_ax,cmap=cmap,norm=norm,extend='both',spacing='uniform')
cmap.set_over('green')
cmap.set_under('green')
这里是数据(异常值为1.69):
Data;A;B;C;D;E;F;G;H;I;J;K
A;1.2;0;0;0;0;1.69;0;0;1.69;1.69;0
B;0;0;0;0;0;1.69;0;0;1.69;1.69;0
C;0;0;0;0;0;1.69;0;0.45;1.69;1.69;0.92
D;1;0;-0.7;-1.2;0;1.69;0;0;1.69;1.69;0
E;0;0;0;0;0;1.69;0;0;1.69;1.69;0
F;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69
G;0;0;0;0;0;1.69;0;0;1.69;1.69;0
H;0;0;0;0;0;1.69;0;0;1.69;1.69;0
I;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69
J;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69;1.69
K;0;0;0;0;0;1.69;0;0;1.69;1.69;0
感激任何帮助