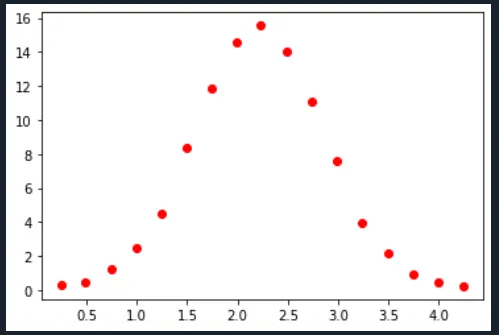
大家好,我想从实验结果中获取一组数据的正态拟合。由于我刚开始学习Python,不知道从哪里开始。这是我的实验数据,它是一个粒径分布。我想获得平均值和标准差。x表示大小,y表示频率。
非常感谢您提前的帮助!
import matplotlib.pyplot as plt
import numpy as np
x=([0.251839516,0.490440575,0.744647994,0.990643452,1.244142316,1.488611658,1.741274792,1.986416351,2.232538986,2.495993944,2.736393641,2.985059803,3.241792581,3.497435276,3.744829674,3.991788039,4.23860106])
y=([0.271164269,0.492366389,1.256781226,2.468772142,4.479769871,8.376708554,11.85803482,14.57231794,15.56056321,14.05547313,11.11227252,7.625604845,3.947070401,2.186355791,0.937144587,0.455061317,0.228687358])
plt.scatter(x,y,color='red',label='Experiment')