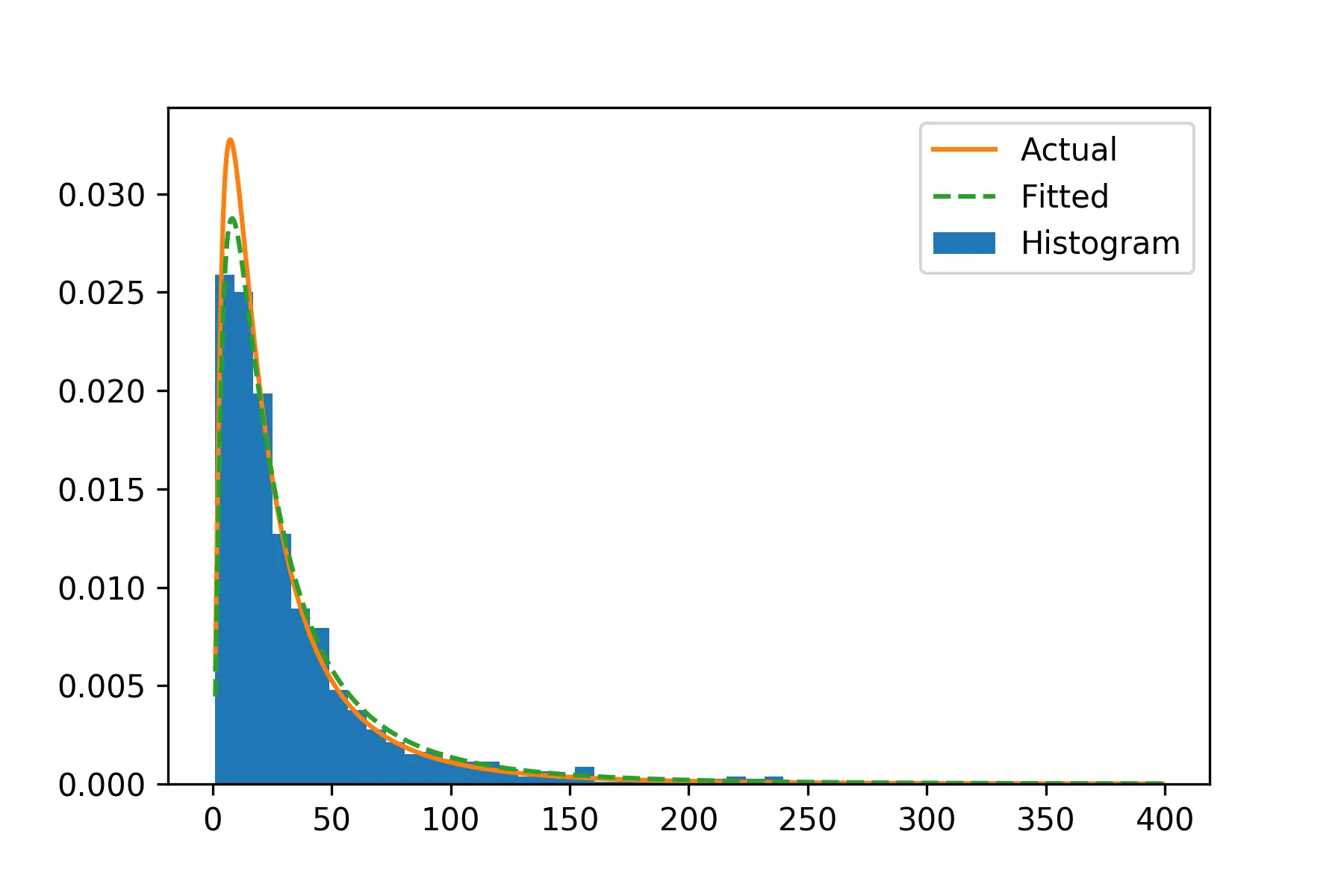
我有一组按体积分数百分比排列的粒径分布数据,如下所示:
size %
6.68 0.05
9.92 1.15
etc.
我需要将这些数据拟合成对数正态分布,计划使用Python的 stats.lognorm.fit 函数来实现,但根据我所阅读到的,这似乎需要将输入作为一个变量数组而不是分组数据。
我计划使用for循环遍历数据,并将每个大小条目.extend到一个占位符数组中,以创建与分组数据相对应的变量列表数组。
尽管如此,这似乎非常丑陋和低效,可能有一种简单的方法可以解决。是否有一种方法可以将分组数据输入到stats.lognorm.fit函数中?