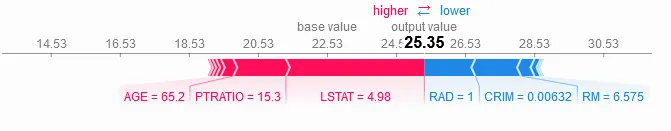
我想分析使用
tidymodels包拟合的我的树集合,并生成SHAP值图,例如单个观察的图表。
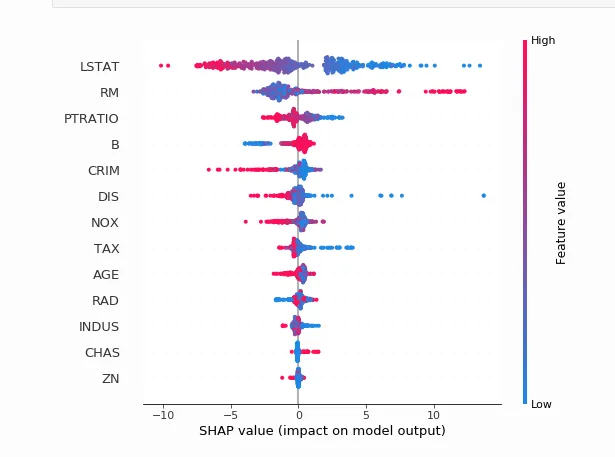
总结一下我的数据集所有特征的影响,例如:
DALEXtra提供了一个函数,用于为tidymodels explain.tidymodels()创建SHAP值。来自fastshap包的force_plot提供了一个包装器,用于底层python包SHAP的绘图函数。但我不知道如何使该函数与explain.tidymodels()函数的输出配合使用。
问题:如何使用tidymodels和explain.tidymodels在R中生成这样的SHAP图?
MWE(对于使用explain.tidymodels的SHAP值)
library(MASS)
library(tidyverse)
library(tidymodels)
library(parsnip)
library(treesnip)
library(catboost)
library(fastshap)
library(DALEXtra)
set.seed(1337)
rec <- recipe(crim ~ ., data = Boston)
split <- initial_split(Boston)
train_data <- training(split)
test_data <- testing(split) %>% dplyr::select(-crim) %>% as.matrix()
model_default<-
parsnip::boost_tree(
mode = "regression"
) %>%
set_engine(engine = 'catboost', loss_function = 'RMSE')
#sometimes catboost is not loaded correctly the following two lines
#ensure prevent fitting errors
#https://github.com/curso-r/treesnip/issues/21 error is mentioned on last post
set_dependency("boost_tree", eng = "catboost", "catboost")
set_dependency("boost_tree", eng = "catboost", "treesnip")
model_fit_wf <- model_fit_wf <- workflow() %>% add_model(model_tune) %>% add_recipe(rec) %>% {parsnip::fit(object = ., data = train_data)}
SHAP_wf <- explain_tidymodels(model_fit_wf, data = X, y = train_data$crim, new_data = test_data
extract_fit_engine()和bake()。 - Julia Silge