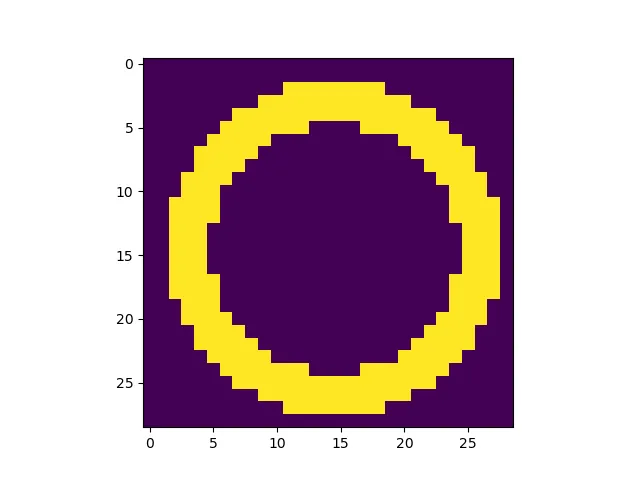
I have the following code:
import numpy as np
import matplotlib.pyplot as plt
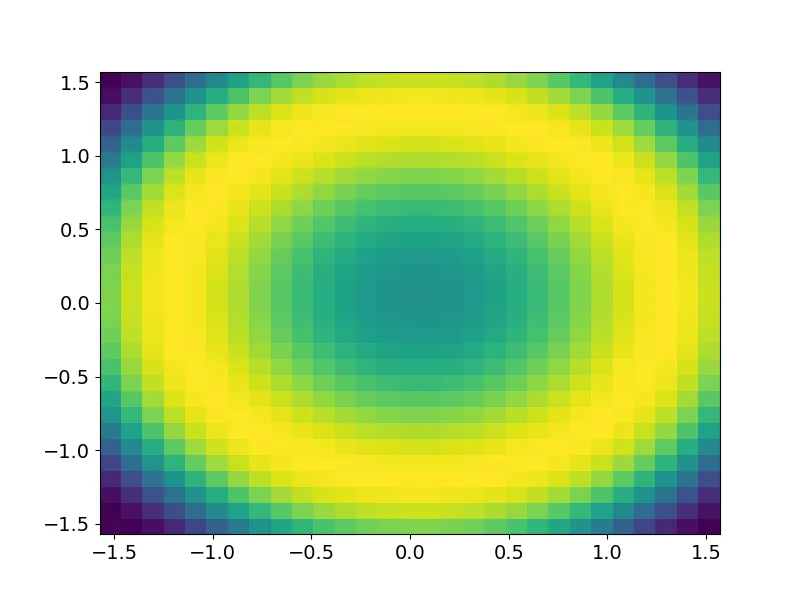
x = np.linspace(-np.pi/2, np.pi/2, 30)
y = np.linspace(-np.pi/2, np.pi/2, 30)
x,y = np.meshgrid(x,y)
z = np.sin(x**2+y**2)[:-1,:-1]
fig,ax = plt.subplots()
ax.pcolormesh(x,y,z)
现在假设我想突出显示某些网格框的边缘:
highlight = (z > 0.9)
我可以使用轮廓函数,但这会导致“平滑”的轮廓。我只想突出显示区域的边缘,沿着网格框的边缘。
我最接近的方法是添加类似以下内容:
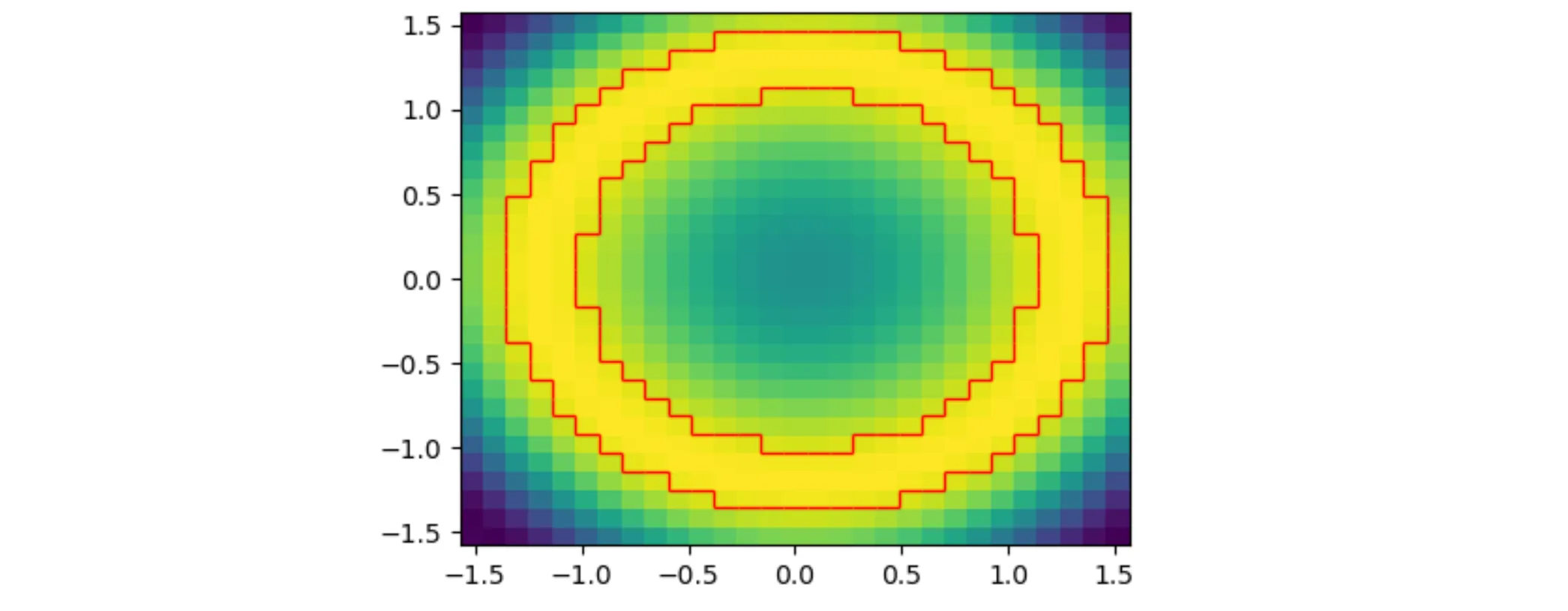
highlight = np.ma.masked_less(highlight, 1)
ax.pcolormesh(x, y, highlight, facecolor = 'None', edgecolors = 'w')
这给出了这个图形:
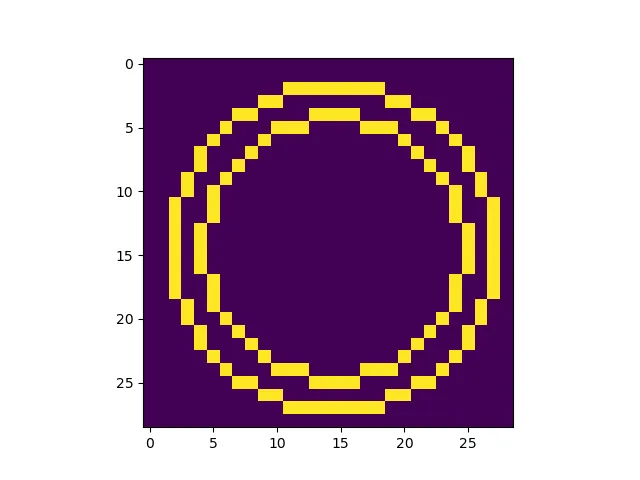
 这很接近,但我真正想要的是只突出显示“甜甜圈”的外边缘和内边缘。
这很接近,但我真正想要的是只突出显示“甜甜圈”的外边缘和内边缘。因此,我正在寻找一些轮廓和pcolormesh函数的混合体 - 跟随某个值的轮廓,但是步进网格单元而不是点对点连接。这有意义吗?
附注:在pcolormesh参数中,我有
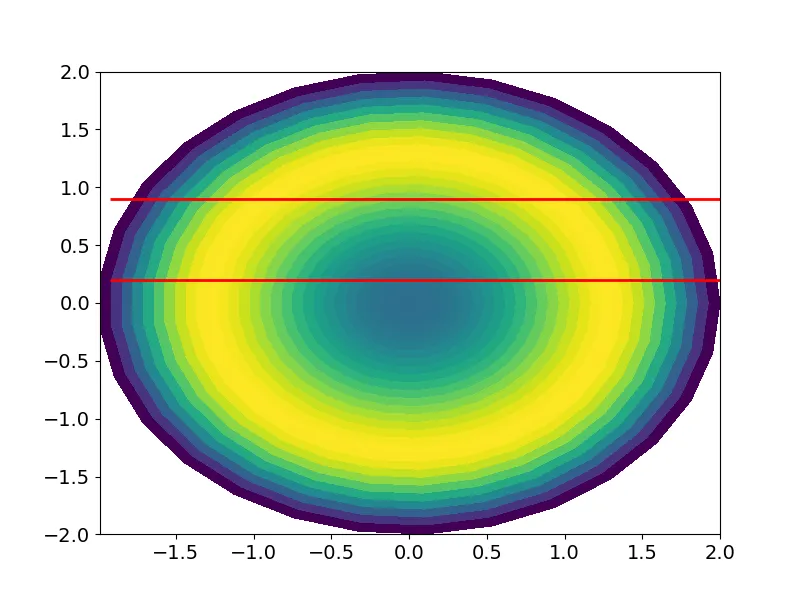
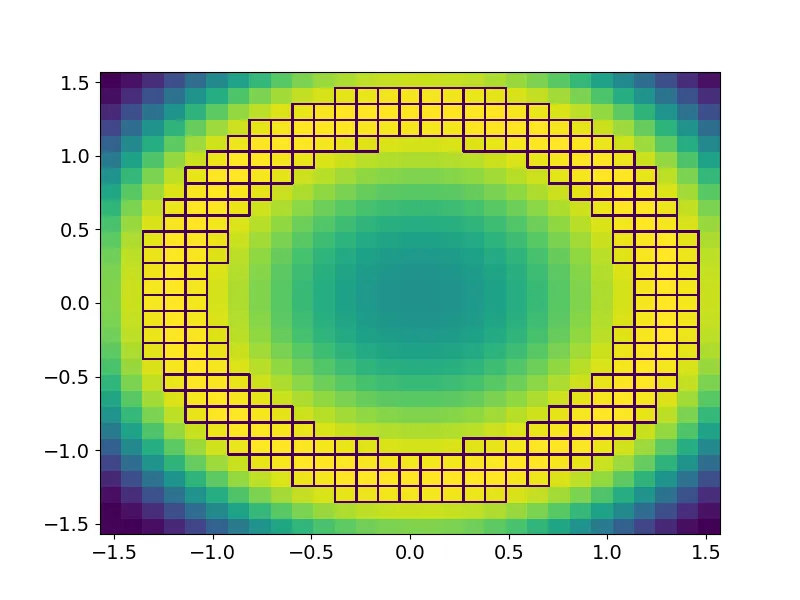
edgecolors='w',但边缘仍然变为蓝色。发生了什么事?编辑:JohanC最初的答案使用add_iso_line()适用于所提出的问题。然而,我使用的实际数据是非常不规则的x,y网格,不能转换为1D(如
add_iso_line()所需)。我正在使用已从极坐标(rho,phi)转换为笛卡尔(x,y)的数据。 JohanC提出的2D解决方案似乎不适用于以下情况:
import numpy as np
import matplotlib.pyplot as plt
from scipy import ndimage
def pol2cart(rho, phi):
x = rho * np.cos(phi)
y = rho * np.sin(phi)
return(x, y)
phi = np.linspace(0,2*np.pi,30)
rho = np.linspace(0,2,30)
pp, rr = np.meshgrid(phi,rho)
xx,yy = pol2cart(rr, pp)
z = np.sin(xx**2 + yy**2)
scale = 5
zz = ndimage.zoom(z, scale, order=0)
fig,ax = plt.subplots()
ax.pcolormesh(xx,yy,z[:-1, :-1])
xlim = ax.get_xlim()
ylim = ax.get_ylim()
xmin, xmax = xx.min(), xx.max()
ymin, ymax = yy.min(), yy.max()
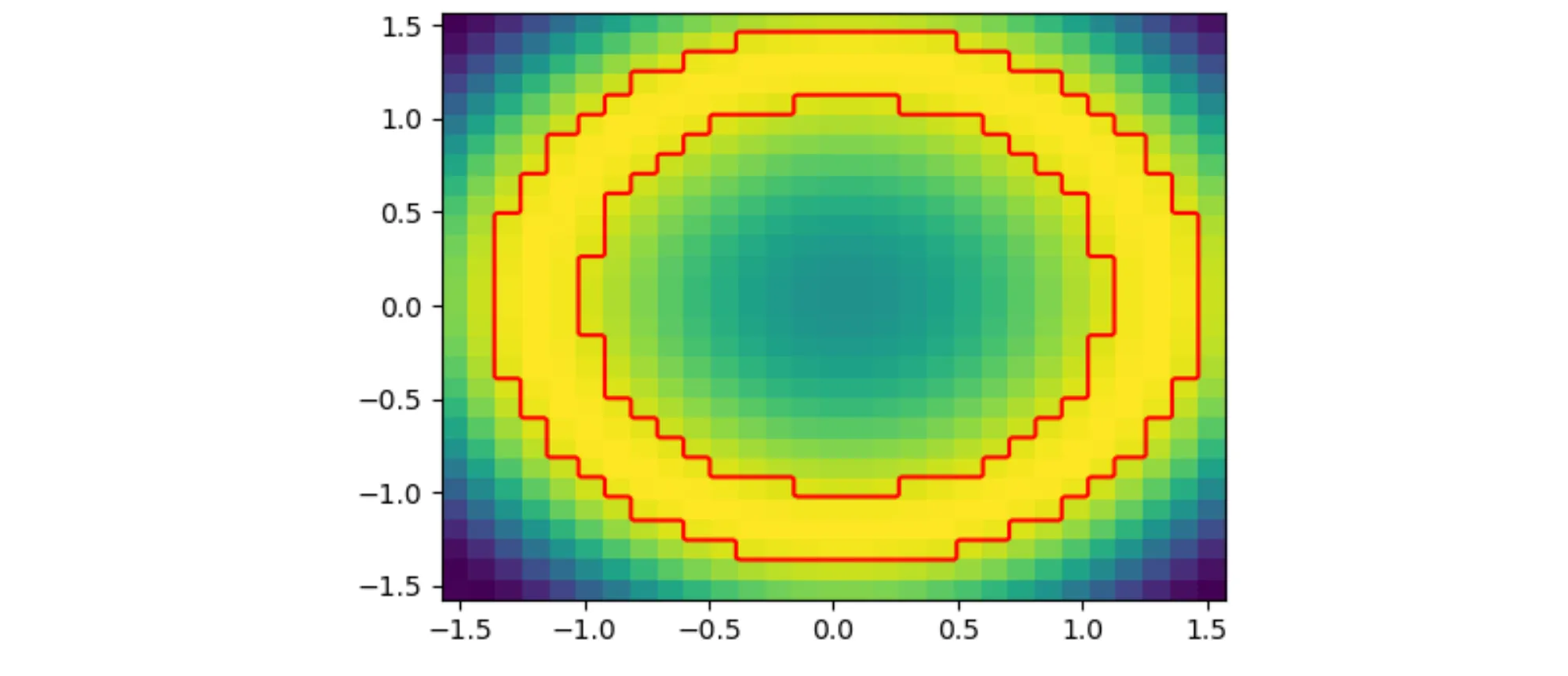
ax.contour(np.linspace(xmin,xmax, zz.shape[1]) + (xmax-xmin)/z.shape[1]/2,
np.linspace(ymin,ymax, zz.shape[0]) + (ymax-ymin)/z.shape[0]/2,
np.where(zz < 0.9, 0, 1), levels=[0.5], colors='red')
ax.set_xlim(*xlim)
ax.set_ylim(*ylim)