我希望将骨头区域进行二值化(使骨头区域为255,其他区域为0),但灰度级分布不够简单(下半部分较亮),无法找到一个阈值直接进行处理。因此,我认为如果能检测其完整轮廓并填充轮廓内的空隙可能是更容易的方法。
原始图像:
我还需要处理两个骨头重叠在一起的问题。(我很抱歉一开始没有提到这个..)
我正在考虑如何分离一对重叠在一起的骨头: http://i.imgur.com/dI5s11L.png
原始图像:
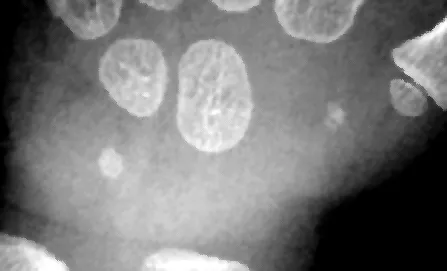
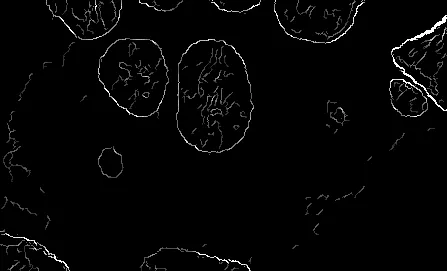
我还需要处理两个骨头重叠在一起的问题。(我很抱歉一开始没有提到这个..)
我正在考虑如何分离一对重叠在一起的骨头: http://i.imgur.com/dI5s11L.png
