我希望进行二元正态分布的离散逼近。也就是说,我希望计算一个矩阵,其中每个条目都是掉落到下图中小方块内的概率。
以下是我目前做的内容:
以下是我目前做的内容:
library(mvtnorm)
library(graphics)
euclide = function(x,y){sqrt(x^2+y^2)}
maxdist = 40
sigma = diag(2)
m = matrix(0,ncol=maxdist*2 + 1, nrow=maxdist*2 + 1)
for (row in -maxdist:maxdist){
for (col in -maxdist:maxdist){
if ( euclide(abs(row), abs(col)) < maxdist ){
lower = c(row-0.5, col-0.5)
upper = c(row+0.5, col+0.5)
p = pmvnorm(lower = lower , upper = upper, mean = c(0,0), sigma = sigma)
} else {
p = 0
}
m[row + maxdist + 1,col + maxdist + 1] = p
}
}
m = m[rowSums(m)!=0,colSums(m)!=0]
contour(m, levels = exp(-20:0), xlim=c(0.3,0.7), ylim=c(0.3,0.7))
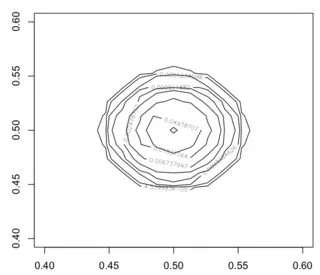
这个程序可以正常工作。但是当maxdist较大时,速度会比较慢。我希望能够提高计算速度,但这不是我的主要问题...
我的主要问题是,使用我的方法时,我无法改变靠近中心的小方块数量,以便更好地逼近平均值。我只能在周围添加方块。换句话说,我想能够设置双变量正态分布的两个轴的方差。
N(0,1)的随机数生成联合正态变量?(例如,这里的幻灯片269-272页)或者你是为了其他特定目的而进行这个练习吗? - MichaelChirico