更新 2:
在我看来,@piRSquared 的 numpy 解决方案是最常见的情况下的赢家。以下是他的答案,只进行了最少的修改以将列分配给原始数据帧(这是我在所有测试中都做的,以保持与原始问题示例的一致性)。
col_mask = df.columns.str.startswith('option')
options = df.columns[col_mask]
v = np.column_stack([*map(df.get, options)])
df.assign(min_value = np.nanmin(v, axis=1),
min_column = options[np.nanargmin(v, axis=1)])
如果你有很多列(超过10000列),你应该小心谨慎,因为在这些极端情况下,结果可能会显著改变。
更新1:
根据我的测试,单独调用min和idxmin是基于所有建议答案中最快的方法。
尽管不是同时进行(请参见下面的直接答案),但最好使用DataFrame.lookup在列索引(min_column列)上,以避免搜索值(min_values)。
因此,您只需遍历结果min_column系列 - 这是O(n),而不是遍历整个矩阵 - 这是O(n*m):
df = pd.DataFrame({
'id': [0,1,2,3,4],
'option_1': [10, np.nan, np.nan, 400, 600],
'option_2': [np.nan, 20, 300, np.nan, 700],
'option_3': [np.nan, 200, 30, np.nan, 50],
'option_4': [110, np.nan, np.nan, 40, 50],
})
df['min_column'] = df.filter(like='option').idxmin(1)
df['min_value'] = df.lookup(df.index, df['min_column'])
直接回答(不够高效)
由于您询问了如何“在同一次调用中计算值”(假设是因为您为问题简化了示例),您可以尝试使用lambda表达式:
def min_idxmin(x):
_idx = x.idxmin()
return _idx, x[_idx]
df['min_column'], df['min_value'] = zip(*df.filter(like='option').apply(
lambda x: min_idxmin(x), axis=1))
要明确的是,虽然这里删除了第二个搜索(用
x[_idx]直接访问),但这很可能需要更长的时间,因为您没有利用pandas/numpy的向量化属性。
总之,pandas/numpy向量化操作非常快速。
摘要总结:
使用df.lookup似乎没有任何优势,单独调用min和idxmin比使用查找更好,这是令人惊叹的,并值得自己提出问题。
时间摘要:
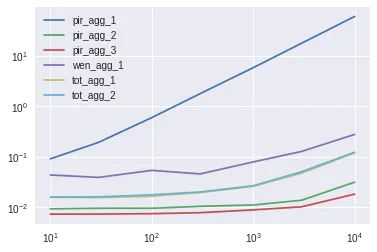
我测试了一个具有10000行和10列的数据框(初始示例中的option_序列)。由于我得到了一些意外的结果,所以我还进行了1000x1000和100x10000的测试。根据结果:
使用 numpy,就像@piRSquared(test8)建议的一样是最好的选择,只有在列数很多时(100,10000),它开始表现较差,但这并不能证明通常使用numpy不好。Test9试图在numpy中使用索引,但总体上表现更差。
分别调用min和idxmin对于10000x10的情况表现最佳,甚至好于Dataframe.lookup(虽然在100x10000的情况下,Dataframe.lookup的结果表现更好)。尽管数据的形状影响结果,但我认为有10000列有点不切实际。
@Wen提供的解决方案在性能上跟进,虽然它不比分别调用idxmin和min或使用Dataframe.lookup更好。我进行了额外的测试(见test7()),因为我觉得操作(reset_index和zip)的添加可能会干扰结果。即使它不执行赋值(我无法想出如何使用head(1)进行赋值),它仍比test1和test2差。@Wen,您能帮我吗?
@Wen的解决方案在有更多列时(1000x1000或100x10000),性能会下降,这是有道理的,因为排序比搜索慢。在这种情况下,我建议使用的lambda表达式表现更好。
任何其他使用lambda表达式或使用转置(T)的解决方案都落后于前面提到的几种方法。我建议使用的lambda表达式需要大约1秒钟,比@piRSquared和@RafaelC建议的transpose T所需的~11秒快得多。
使用10000行x 10列的TimeIt结果(pandas 0.23.4):
使用以下10000行和10列的数据框:
import pandas as pd
import numpy as np
df = pd.DataFrame(np.random.randint(0,100,size=(10000, 10)), columns=[f'option_{x}' for x in range(1,11)]).reset_index()
Calling the two columns twice separatedly:
def test1():
df['min_column'] = df.filter(like='option').idxmin(1)
df['min_value'] = df.filter(like='option').min(1)
13 ms ± 580 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
Calling the lookup (it is slower for this case!):
def test2():
df['min_column'] = df.filter(like='option').idxmin(1)
df['min_value'] = df.lookup(df.index, df['min_column'])
%timeit -n 100 test2()
Using apply and min_idxmin(x):
def min_idxmin(x):
_idx = x.idxmin()
return _idx, x[_idx]
def test3():
df['min_column'], df['min_value'] = zip(*df.filter(like='option').apply(
lambda x: min_idxmin(x), axis=1))
%timeit -n 10 test3()
Using agg['min', 'idxmin'] by @piRSquared:
def test4():
df['min_column'], df['min_value'] = zip(*df.set_index('index').filter(like='option').T.agg(['min', 'idxmin']).T.values)
%timeit -n 1 test4()
Using agg['min', 'idxmin'] by @RafaelC:
def test5():
df['min_column'], df['min_value'] = zip(*df.filter(like='option').agg(lambda x: x.agg(['min', 'idxmin']), axis=1).values)
%timeit -n 1 test5()
Sorting values by @Wen:
def test6():
df['min_column'], df['min_value'] = zip(*df.filter(like='option').stack().sort_values().groupby(level=[0]).head(1).reset_index(level=1).values)
%timeit -n 100 test6()
Sorting values by @Wen modified by me to make the comparison fairer due to overload of assigment operation (I explained why in the summary at the beginning):
def test7():
df.filter(like='option').stack().sort_values().groupby(level=[0]).head(1)
%timeit -n 100 test7()
Using numpy:
def test8():
col_mask = df.columns.str.startswith('option')
options = df.columns[col_mask]
v = np.column_stack([*map(df.get, options)])
df.assign(min_value = np.nanmin(v, axis=1),
min_column = options[np.nanargmin(v, axis=1)])
%timeit -n 100 test8()
Using numpy but avoid the search (indexing instead):
def test9():
col_mask = df.columns.str.startswith('option')
options = df.columns[col_mask]
v = np.column_stack([*map(df.get, options)])
idxmin = np.nanargmin(v, axis=1)
df.assign(min_value = v[range(v.shape[0]), idxmin],
min_column = options[idxmin])
%timeit -n 100 test9()
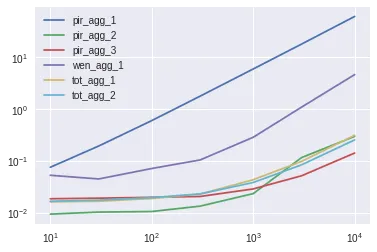
1000行x1000列的TimeIt结果:
我进行了更多的测试,使用了1000x1000的形状:
df = pd.DataFrame(np.random.randint(0,100,size=(1000, 1000)), columns=[f'option_{x}' for x in range(1,1001)]).reset_index()
尽管结果会发生改变:
test1 ~27.6ms
test2 ~29.4ms
test3 ~135ms
test4 ~1.18s
test5 ~1.29s
test6 ~287ms
test7 ~290ms
test8 ~25.7
test9 ~26.1
100行 x 10000列的TimeIt结果:
我进行了一个100x10000的形状的更多测试:
df = pd.DataFrame(np.random.randint(0,100,size=(100, 10000)), columns=[f'option_{x}' for x in range(1,10001)]).reset_index()
虽然结果会改变:
test1 ~46.8ms
test2 ~25.6ms
test3 ~101ms
test4 ~289ms
test5 ~276ms
test6 ~349ms
test7 ~301ms
test8 ~121ms
test9 ~122ms
O(1),搜索为O(n)。实际上,直接访问的k似乎非常大,矢量化搜索非常快。我假设直接访问也应该被矢量化了(我错了吗?),在这种情况下,我们比较的是矢量化的搜索和直接访问,再次强调,我的假设可能是错误的。 - toto_tico.lookup()比[]/.iloc()慢,因为它需要标签(而不是索引)。正如你的答案所示,列越多,速度越慢。此外,如果您说明您关心的典型或最大维度范围:10,000列或更多?1,000行或更多?矩阵是否稀疏,您是否关心NaN条目,NaN是否可以稀疏表示?请参见pandas Sparse data structures。 - smci