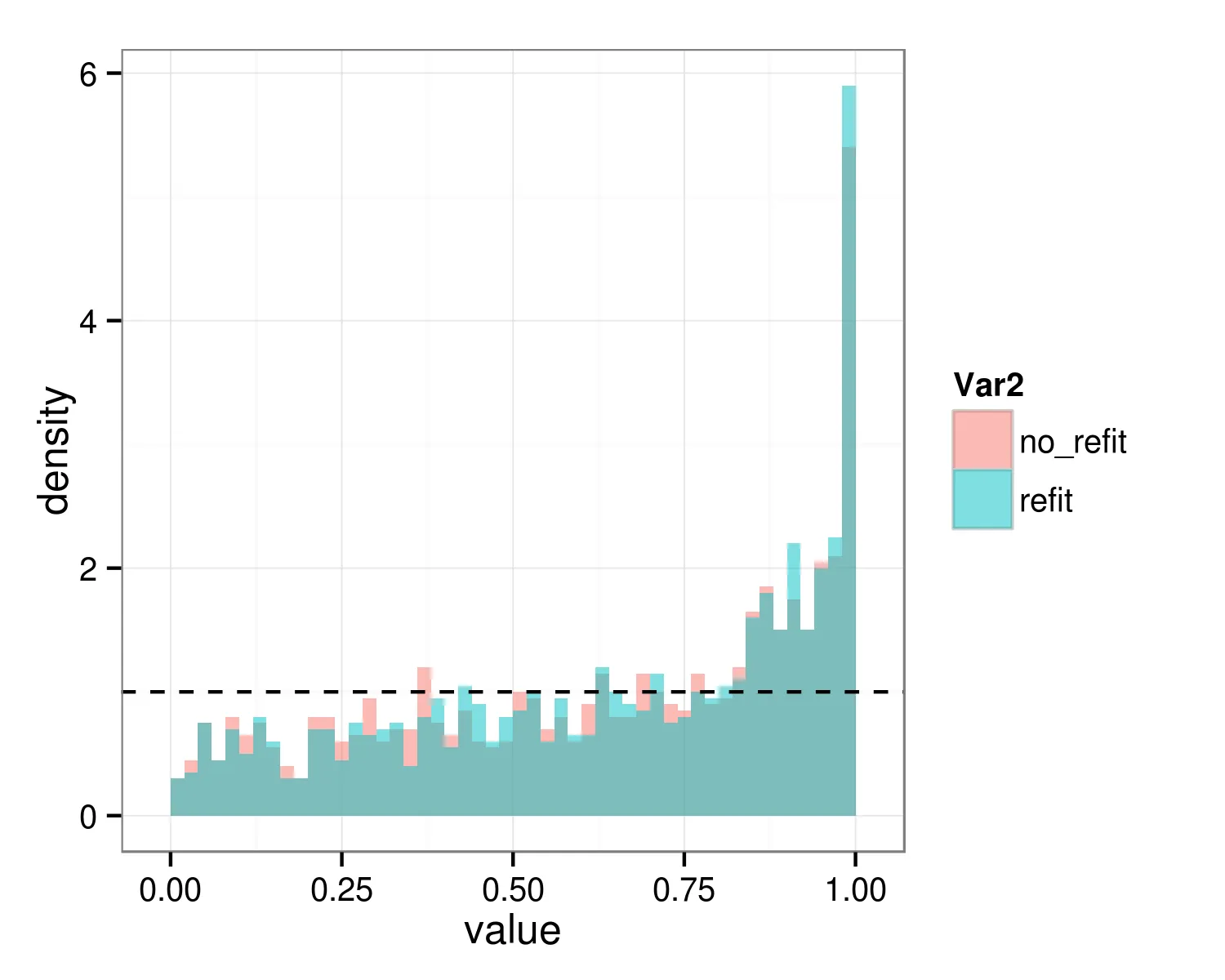
我目前在测试是否应该在我的lmer模型中包含某些随机效应。为此,我使用anova函数。到目前为止,我的步骤是使用lmer()函数调用拟合模型,并使用REML=TRUE(默认选项)。然后,在其中一个模型上调用anova(),该模型包括要测试的随机效应,而另一个模型则不包括。然而,众所周知,anova()函数会重新拟合ML模型,但是在新版本的anova()中,您可以通过设置选项refit=FALSE来防止anova()这样做。为了测试随机效应,我应该在对anova()的调用中设置refit=FALSE还是不设置?(如果我设置refit=FALSE,则p值往往会更低。当我设置refit=FALSE时,p值是否反保守?)
方法1:
mod0_reml <- lmer(x ~ y + z + (1 | w), data=dat)
mod1_reml <- lmer(x ~ y + z + (y | w), data=dat)
anova(mod0_reml, mod1_reml)
这将导致anova()使用ML而不是REML重新拟合模型。(较新版本的anova()函数也会输出有关此信息的信息。)
方法2:
mod0_reml <- lmer(x ~ y + z + (1 | w), data=dat)
mod1_reml <- lmer(x ~ y + z + (y | w), data=dat)
anova(mod0_reml, mod1_reml, refit=FALSE)
这将导致anova()在原始模型上执行其计算,即使用REML=TRUE。
哪种方法是正确的,以测试是否应该包括随机效应?
感谢任何帮助。