我希望在
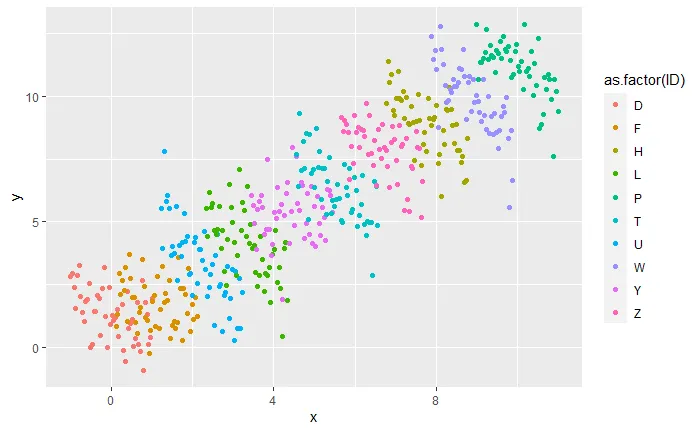
这是我的工作示例:我定义了一个带有辛普森悖论的数据集:全局呈上升趋势,但每个组内呈下降趋势。
你可以看到类似于Simpson悖论的维基页面示例的内容:
现在我使用
但是当我想要使用随机效果时:
R中使用h2o进行glm回归,但需要使用随机效应(HGLM,似乎可以从this page实现)。但我还没有成功地使它工作,并且遇到了我不理解的错误。这是我的工作示例:我定义了一个带有辛普森悖论的数据集:全局呈上升趋势,但每个组内呈下降趋势。
library(tidyverse)
library(ggplot2)
library(h2o)
library(data.table)
global_slope <- 1
global_int <- 1
Npoints_per_group <- 50
N_groups <- 10
pentes <- rnorm(N_groups,-1,.5)
centers_x <- seq(0,10,length = N_groups)
center_y <- global_slope*centers_x + global_int
group_spread <- 2
group_names <- sample(LETTERS,N_groups)
df <- lapply(1:N_groups,function(i){
x <- seq(centers_x[i]-group_spread/2,centers_x[i]+group_spread/2,length = Npoints_per_group)
y <- pentes[i]*(x- centers_x[i])+center_y[i]+rnorm(Npoints_per_group)
data.table(x = x,y = y,ID = group_names[i])
}) %>% rbindlist()
你可以看到类似于Simpson悖论的维基页面示例的内容:
ggplot(df,aes(x,y,color = as.factor(ID)))+
geom_point()
lm(y~x,data = df) %>%
summary()
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.28187 0.13077 9.803 <2e-16 ***
x 0.94147 0.02194 42.917 <2e-16 ***
标准的多层回归应该是这样的:
library(lme4)
library(lmerTest)
lmer( y ~ x + (1+x|ID) ,data = df) %>%
summary()
并且会正确估计下降趋势:
Fixed effects:
Estimate Std. Error df t value Pr(>|t|)
(Intercept) 11.7192 2.6218 8.8220 4.470 0.001634 **
x -1.0418 0.1959 8.9808 -5.318 0.000486 ***
现在我使用
h2o 进行测试:library(h2o)
h2o.init()
df2 <- as.h2o(df)
test_glm <- h2o.glm(family = "gaussian",
x = "x",
y = "y",
training_frame = df2,
lambda = 0,
compute_p_values = TRUE)
test_glm
它表现良好,类似于上面的线性模型:
Coefficients: glm coefficients
names coefficients std_error z_value p_value standardized_coefficients
1 Intercept 1.281868 0.130766 9.802785 0.000000 5.989232
2 x 0.941473 0.021937 42.916536 0.000000 3.058444
但是当我想要使用随机效果时:
test_glm2 <- h2o.glm(family = "gaussian",
x = "x",
y = "y",
training_frame = df2,
random_columns = "ID",
lambda = 0,
compute_p_values = TRUE)
我收到了以下内容:
在函数 .h2o.checkAndUnifyModelParameters(algo = algo, allParams = ALL_PARAMS) 中出错:向量 random_columns 必须是数字类型,但得到的却是字符。
即使我强制将 df2$ID <- as.numeric(df2$ID)。
我做错了什么?使用 lmer 找到类似于混合效应模型(即随机斜率和截距)的适当方法是什么?
编辑
我按照Erin LeDell的建议,改用列号。现在出现了一个我不理解的不同错误:
df2$ID <- as.factor(df2$ID)
test_glm2 <- h2o.glm(family = "gaussian",
x = "x",
y = "y",
training_frame = df2,
random_columns = c(3),
HGLM = TRUE,
lambda = 0,
compute_p_values = TRUE)
DistributedException from localhost/127.0.0.1:54321: 'null', caused by java.lang.NullPointerException
DistributedException from localhost/127.0.0.1:54321: 'null', caused by java.lang.NullPointerException
at water.MRTask.getResult(MRTask.java:660)
at water.MRTask.getResult(MRTask.java:670)
at water.MRTask.doAll(MRTask.java:530)
at water.MRTask.doAll(MRTask.java:482)
at hex.glm.GLM$GLMDriver.fitCoeffs(GLM.java:1334)
at hex.glm.GLM$GLMDriver.fitHGLM(GLM.java:1505)
at hex.glm.GLM$GLMDriver.fitModel(GLM.java:2060)
at hex.glm.GLM$GLMDriver.computeSubmodel(GLM.java:2526)
at hex.glm.GLM$GLMDriver.doCompute(GLM.java:2664)
at hex.glm.GLM$GLMDriver.computeImpl(GLM.java:2561)
at hex.ModelBuilder$Driver.compute2(ModelBuilder.java:247)
at hex.glm.GLM$GLMDriver.compute2(GLM.java:1188)
at water.H2O$H2OCountedCompleter.compute(H2O.java:1658)
at jsr166y.CountedCompleter.exec(CountedCompleter.java:468)
at jsr166y.ForkJoinTask.doExec(ForkJoinTask.java:263)
at jsr166y.ForkJoinPool$WorkQueue.runTask(ForkJoinPool.java:976)
at jsr166y.ForkJoinPool.runWorker(ForkJoinPool.java:1479)
at jsr166y.ForkJoinWorkerThread.run(ForkJoinWorkerThread.java:104)
编辑2:
我实际上找到了一种方法来消除上述错误,方法是添加
rand_link = c("identity"),
rand_family = c("gaussian"),
对于h2o.glm的参数:
h2o.glm(family = "gaussian",
rand_link = c("identity"),
rand_family = c("gaussian"),
# compute_p_values = TRUE,
x = "x",
y = "y",
training_frame = df2,
random_columns = c(3),
HGLM = TRUE,
lambda = 0)
程序正常运行。但是当我设置 compute_p_values = TRUE 后,出现了一个新的错误:
Error in .h2o.doSafeREST(h2oRestApiVersion = h2oRestApiVersion, urlSuffix = page, :
ERROR MESSAGE:
degrees of freedom (0)
Error: DistributedException from localhost/127.0.0.1:54321: 'null', caused by java.lang.NullPointerException- denis