使用不同的模型对进行配对,并基于重复样本的分类准确性比较模型。可以轻松扩展到其他指标。
library(dplyr)
library(tibble)
library(ggplot2)
library(tidyr)
library(rsample)
library(recipes)
library(parsnip)
library(workflows)
library(tune)
library(yardstick)
library(workflowsets)
set.seed(123)
iris <- iris %>% mutate(
is_versicolor = ifelse(Species == "versicolor", "versicolor", "not_versicolor")) %>%
mutate(is_versicolor = factor(is_versicolor, levels = c("versicolor", "not_versicolor")))
iris_split <- initial_split(iris, strata = is_versicolor, prop = 0.8)
iris_train <- training(iris_split)
iris_test <- testing(iris_split)
rec_normal <- recipe(is_versicolor ~ Sepal.Width, data = iris_train)
rec_alternative <- recipe(is_versicolor ~ Sepal.Length, data = iris_train)
iris_model <- rand_forest() %>% set_engine("ranger") %>% set_mode("classification")
set.seed(234)
iris_folds <- vfold_cv(iris_train)
iris_folds
iris_set <-
workflow_set(
list(rec_normal, rec_alternative),
list(iris_model),
cross = TRUE
)
doParallel::registerDoParallel()
set.seed(2021)
iris_rs <-
workflow_map(
iris_set,
"fit_resamples",
resamples = iris_folds
)
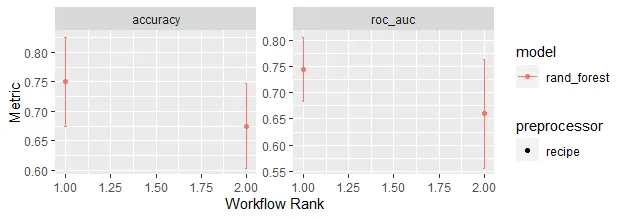
autoplot(iris_rs)
# Extract resample accuracies
model_1_rs <- iris_rs[1,][[4]][[1]]$.metrics
model_2_rs <- iris_rs[2,][[4]][[1]]$.metrics
model_acc <- tibble(model_1 = NA, model_2 = NA)
for (i in 1:10) {
model_acc[i, 1] <- model_1_rs[[i]][[".estimate"]][1]
model_acc[i, 2] <- model_2_rs[[i]][[".estimate"]][1]
}
model_acc <- model_acc |> pivot_longer(cols = everything(), names_to = "model", values_to = "acc")
aov_results <- aov(acc ~ model, data = model_acc)
summary(aov_results)
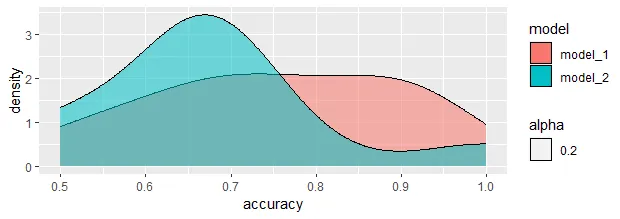
ggplot(data = model_acc, aes(fill = model)) +
geom_density(aes(x = acc, alpha = 0.2)) +
labs(x = "accuracy")
给出 p 值:
> summary(aov_results)
Df Sum Sq Mean Sq F value Pr(>F)
model 1 0.0281 0.02813 1.378 0.256
Residuals 18 0.3674 0.02041
从不同的角度观察模型准确性的p值:
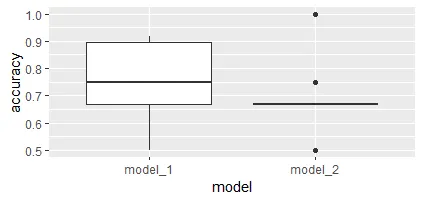
首先可视化变化:
model_acc |> ggplot(aes(x = model, y = acc)) +
geom_boxplot() +
labs(y = 'accuracy')
 然后计算一个测试统计量:
然后计算一个测试统计量:
observed_statistic <- model_acc %>%
specify(acc ~ model) %>%
calculate(stat = "diff in means", order = c("model_1", "model_2"))
observed_statistic
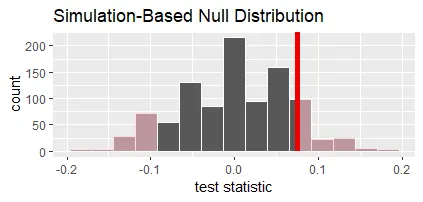
然后进行分布的模拟:
null_dist_2_sample <- model_acc %>%
specify(acc ~ model) %>%
hypothesize(null = "independence") %>%
generate(reps = 1000, type = "permute") %>%
calculate(stat = "diff in means" ,order = c("model_1", "model_2"))
并绘制:
null_dist_2_sample %>%
visualize() +
shade_p_value(observed_statistic,
direction = "two-sided") +
labs(x = "test statistic")
 并获取p值:
并获取p值:
p_value_2_sample <- null_dist_2_sample %>%
get_p_value(obs_stat = observed_statistic,
direction = "two-sided")
p_value_2_sample
# A tibble: 1 × 1
p_value
<dbl>
1 0.228
这与aov的p值几乎相同。
请注意,由于两个模型的准确性接近,p值较高。




lme4::lmer()。查看tune_race_anova()的内部以了解可能的方法。 - Julia Silgeiris_model <- logistic_reg() |> set_engine("glm") |> set_mode("classification")- Isaiah