我想使用 numpy.polyfit 进行物理计算,因此我需要误差的数量级。
2个回答
40
如果在调用
polyfit时指定full=True,它将包括额外的信息:>>> x = np.arange(100)
>>> y = x**2 + 3*x + 5 + np.random.rand(100)
>>> np.polyfit(x, y, 2)
array([ 0.99995888, 3.00221219, 5.56776641])
>>> np.polyfit(x, y, 2, full=True)
(array([ 0.99995888, 3.00221219, 5.56776641]), # coefficients
array([ 7.19260721]), # residuals
3, # rank
array([ 11.87708199, 3.5299267 , 0.52876389]), # singular values
2.2204460492503131e-14) # conditioning threshold
返回的残差值是拟合误差平方和,不确定这是否符合您的要求:
>>> np.sum((np.polyval(np.polyfit(x, y, 2), x) - y)**2)
7.1926072073491056
- Jaime
2
26
正如您在文档中所看到的:
Returns
-------
p : ndarray, shape (M,) or (M, K)
Polynomial coefficients, highest power first.
If `y` was 2-D, the coefficients for `k`-th data set are in ``p[:,k]``.
residuals, rank, singular_values, rcond : present only if `full` = True
Residuals of the least-squares fit, the effective rank of the scaled
Vandermonde coefficient matrix, its singular values, and the specified
value of `rcond`. For more details, see `linalg.lstsq`.
这意味着如果您进行拟合并得到残差如下:
import numpy as np
x = np.arange(10)
y = x**2 -3*x + np.random.random(10)
p, res, _, _, _ = numpy.polyfit(x, y, deg, full=True)
然后,p 是您的拟合参数,res 将是残差,就像上面描述的那样。下划线是因为您不需要保存最后三个参数,所以可以将它们保存在变量 _ 中,您不会使用它们。这是一种惯例,不是必需的。
@Jaime 的回答解释了残差的含义。你可以看看那些平方偏差的函数(其总和为res)。这对于查看未充分拟合的趋势特别有帮助。残差可能很大是由于统计噪声,或者可能是由于系统的拟合较差造成的,例如:
x = np.arange(100)
y = 1000*np.sqrt(x) + x**2 - 10*x + 500*np.random.random(100) - 250
p = np.polyfit(x,y,2) # insufficient degree to include sqrt
yfit = np.polyval(p,x)
figure()
plot(x,y, label='data')
plot(x,yfit, label='fit')
plot(x,yfit-y, label='var')
因此在这张图中,请注意 x=0 附近的拟合不好:
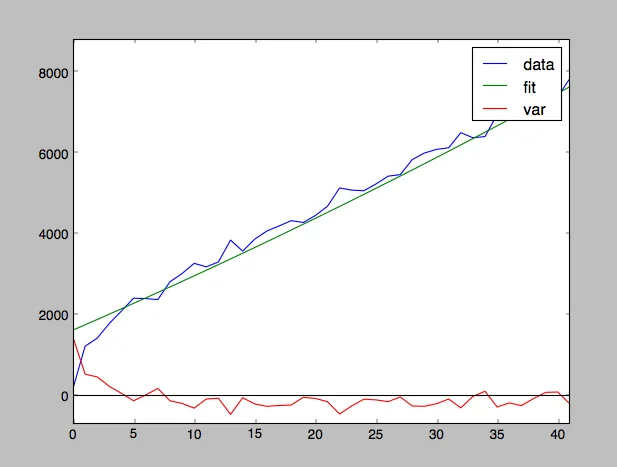
- askewchan
网页内容由stack overflow 提供, 点击上面的可以查看英文原文,
原文链接
原文链接
- 相关问题
- 12 如何使用numpy.polyfit查找斜率和截距的错误
- 6 numpy.polyfit返回空的残差数组
- 7 scipy.curve_fit与numpy.polyfit有不同的协方差矩阵。
- 9 应用numpy.polyfit到xarray数据集
- 3 numpy.polyfit:如何获取估计曲线周围的1-sigma不确定性?
- 5 numpy.polyfit与scipy.odr的区别
- 4 适应参数的numpy.polyfit
- 11 卡方检验 numpy.polyfit (numpy)
- 11 numpy.polyfit与numpy.polynomial.polynomial.polyfit的区别
- 21 numpy.polyfit无法处理NaN值。
np.polyfit使用普通最小二乘算法,使用np.linalg.lstsq,类似于scipy.linalg.lstsq。scipy.odr实现正交最小二乘算法,更精确地说是正交距离回归。 - Cibin Joseph