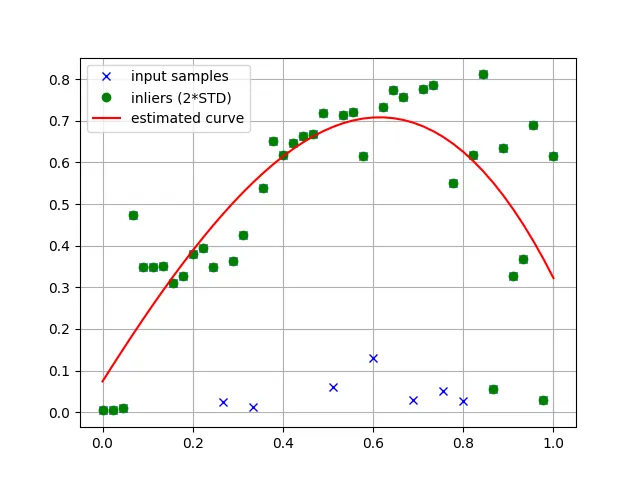
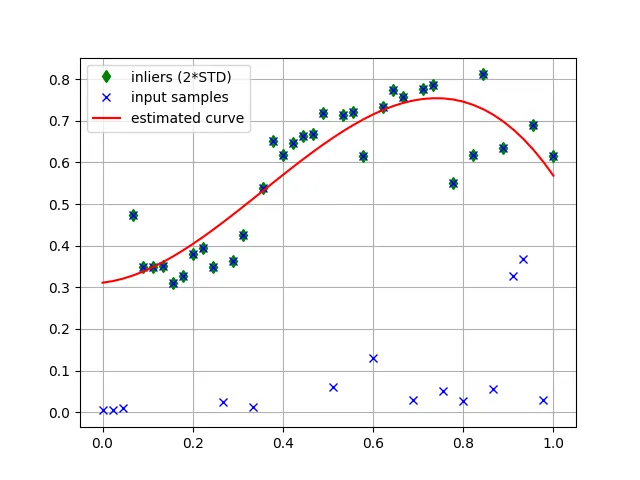
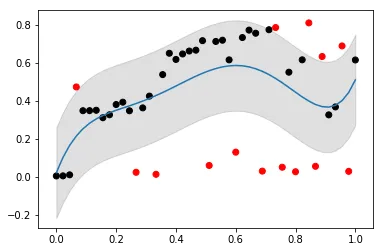
我希望使用以下方法在Python中对数据进行迭代拟合曲线:
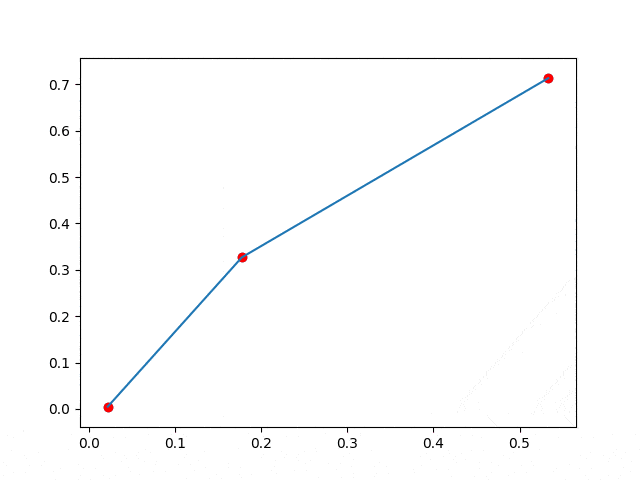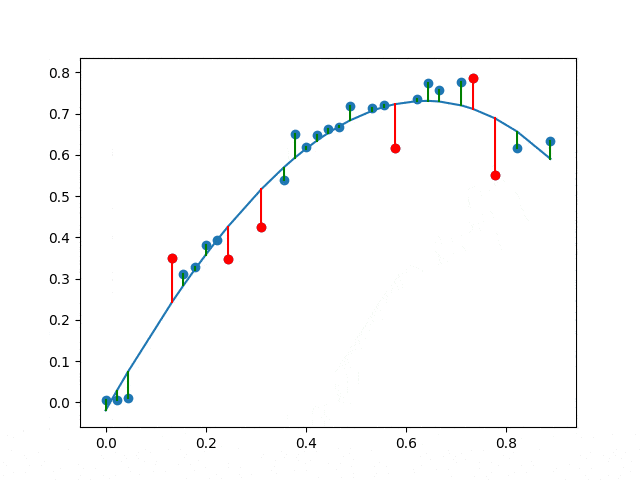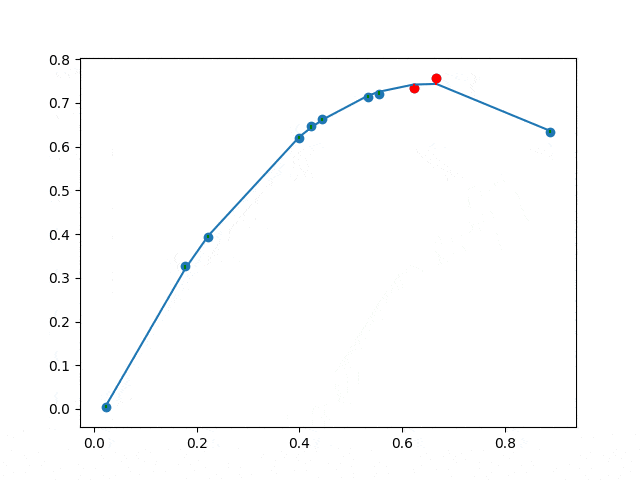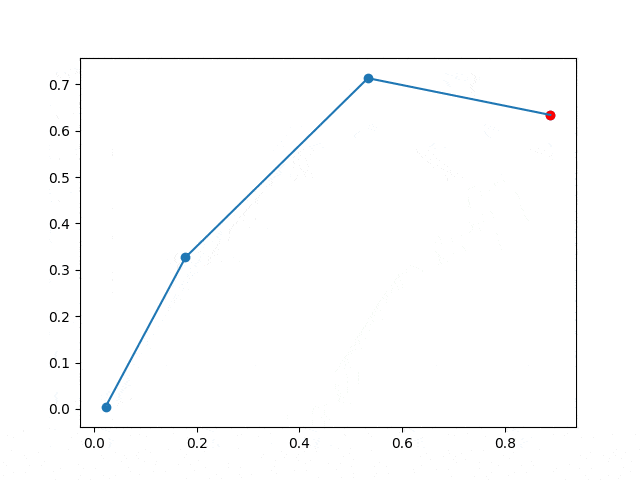
- 拟合多项式曲线(或任何非线性方法)
- 丢弃偏离曲线均值2个标准差以上的值
- 重复步骤1和2,直到所有值都在曲线的置信区间内。
我可以按照以下方式拟合多项式曲线:
vals = array([0.00441025, 0.0049001 , 0.01041189, 0.47368389, 0.34841961,
0.3487533 , 0.35067096, 0.31142986, 0.3268407 , 0.38099566,
0.3933048 , 0.3479948 , 0.02359819, 0.36329588, 0.42535543,
0.01308297, 0.53873956, 0.6511364 , 0.61865282, 0.64750302,
0.6630047 , 0.66744816, 0.71759617, 0.05965622, 0.71335208,
0.71992683, 0.61635697, 0.12985441, 0.73410642, 0.77318621,
0.75675988, 0.03003641, 0.77527201, 0.78673995, 0.05049178,
0.55139476, 0.02665514, 0.61664748, 0.81121749, 0.05521697,
0.63404375, 0.32649395, 0.36828268, 0.68981099, 0.02874863,
0.61574739])
x_values = np.linspace(0, 1, len(vals))
poly_degree = 3
coeffs = np.polyfit(x_values, vals, poly_degree)
poly_eqn = np.poly1d(coeffs)
y_hat = poly_eqn(x_values)
我该如何完成第二步和第三步?