我正在尝试计算带权拓扑重叠(weighted topological overlap)的邻接矩阵,但是我无法使用 numpy 正确完成它。正确实现的 R 函数来自于 WGCNA (https://www.rdocumentation.org/packages/WGCNA/versions/1.67/topics/TOMsimilarity)。计算这个的公式(我认为)在第4个方程式中详细说明,我相信以下是正确的复制。
有谁知道如何正确实现它以反映 WGCNA 版本吗?
是的,我知道 rpy2,但是如果可能的话,我想要使用轻量级的方法。
首先,我的对角线不是 1,并且值与原始值没有一致的误差(例如,不是所有的值都偏离了 x)。
当我在 R 中计算这个时,我使用以下内容:
> library(WGCNA, quiet=TRUE)
> df_adj = read.csv("https://pastebin.com/raw/sbAZQsE6", row.names=1, header=TRUE, check.names=FALSE, sep="\t")
> df_tom = TOMsimilarity(as.matrix(df_adj), TOMType="unsigned", TOMDenom="min")
# ..connectivity..
# ..matrix multiplication (system BLAS)..
# ..normalization..
# ..done.
# I've uploaded it to this url: https://pastebin.com/raw/HT2gBaZC
我不确定我的代码哪里有误。 R 版本的源代码在这里,但它使用了 C 后端脚本,这对我来说非常难以理解。
以下是我在 Python 中的实现:
import pandas as pd
import numpy as np
from sklearn.datasets import load_iris
def get_iris_data():
iris = load_iris()
# Iris dataset
X = pd.DataFrame(iris.data,
index = [*map(lambda x:f"iris_{x}", range(150))],
columns = [*map(lambda x: x.split(" (cm)")[0].replace(" ","_"), iris.feature_names)])
y = pd.Series(iris.target,
index = X.index,
name = "Species")
return X, y
# Get data
X, y = get_iris_data()
# Create an adjacency network
# df_adj = np.abs(X.T.corr()) # I've uploaded this part to this url: https://pastebin.com/raw/sbAZQsE6
df_adj = pd.read_csv("https://pastebin.com/raw/sbAZQsE6", sep="\t", index_col=0)
A_adj = df_adj.values
# Correct TOM from WGCNA for the A_adj
# See above for code
# https://www.rdocumentation.org/packages/WGCNA/versions/1.67/topics/TOMsimilarity
df_tom__wgcna = pd.read_csv("https://pastebin.com/raw/HT2gBaZC", sep="\t", index_col=0)
# My attempt
A = A_adj.copy()
dimensions = A.shape
assert dimensions[0] == dimensions[1]
d = dimensions[0]
# np.fill_diagonal(A, 0)
# Equation (4) from http://dibernardo.tigem.it/files/papers/2008/zhangbin-statappsgeneticsmolbio.pdf
A_tom = np.zeros_like(A)
for i in range(d):
a_iu = A[i]
k_i = a_iu.sum()
for j in range(i+1, d):
a_ju = A[:,j]
k_j = a_ju.sum()
l_ij = np.dot(a_iu, a_ju)
a_ij = A[i,j]
numerator = l_ij + a_ij
denominator = min(k_i, k_j) + 1 - a_ij
w_ij = numerator/denominator
A_tom[i,j] = w_ij
A_tom = (A_tom + A_tom.T)
有一个名为
GTOM的包(https://github.com/benmaier/gtom),但它不适用于加权邻接矩阵。 GTOM的作者也研究了这个问题(使用了更为复杂/高效的NumPy实现,但仍未产生预期结果)。
有人知道如何复制WGCNA实现吗?
编辑:2019年06月20日
我从@scleronomic和@benmaier那里借鉴了一些代码,并在doc string中列出了他们的贡献。该函数可在soothsayer中使用,版本为v2016.06及以上。希望这将使Python中的拓扑重叠比更容易地使用,而不仅限于R。
 https://github.com/jolespin/soothsayer/blob/master/soothsayer/networks/networks.py
https://github.com/jolespin/soothsayer/blob/master/soothsayer/networks/networks.py
import numpy as np
import soothsayer as sy
df_adj = sy.io.read_dataframe("https://pastebin.com/raw/sbAZQsE6")
df_tom = sy.networks.topological_overlap_measure(df_adj)
df_tom__wgcna = sy.io.read_dataframe("https://pastebin.com/raw/HT2gBaZC")
np.allclose(df_tom, df_tom__wgcna)
# True
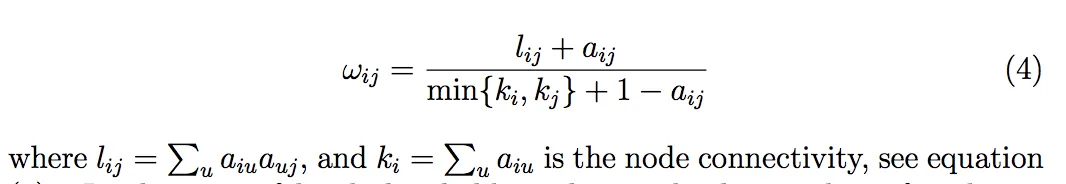
L矩阵的处理有误,哈哈。 - O.rka