是否有适用于 Python 的 SciPy 函数、NumPy 函数或模块可以计算给定特定窗口的一维数组的滑动平均值?
移动平均或滑动平均
2
使用
np.convolve函数:np.convolve(x, np.ones(N)/N, mode='valid')
解释
滑动平均是数学运算卷积的一种情况。对于滑动平均,您沿着输入滑动一个窗口,并计算窗口内容的平均值。对于离散的一维信号,卷积是相同的操作,只是在计算时不是求平均值,而是计算任意线性组合,即将每个元素乘以相应的系数并将结果相加。这些系数,窗口中的每个位置都有一个,有时被称为卷积核。N个值的算术平均值是(x_1 + x_2 + ... + x_N) / N,因此相应的核是(1/N, 1/N, ..., 1/N),通过使用np.ones(N)/N就可以得到这个核。
边缘处理
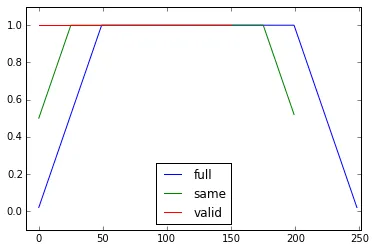
np.convolve函数的mode参数指定如何处理边缘。我选择了valid模式,因为我认为这是大多数人对滑动平均的期望方式,但您可能有其他优先考虑。下面是一个说明不同模式之间差异的图表:
import numpy as np
import matplotlib.pyplot as plt
modes = ['full', 'same', 'valid']
for m in modes:
plt.plot(np.convolve(np.ones(200), np.ones(50)/50, mode=m));
plt.axis([-10, 251, -.1, 1.1]);
plt.legend(modes, loc='lower center');
plt.show()
4
numpy.cumsum,具有更好的复杂度。 - Ulrich Sterndef moving_average(x, N, fill=True):
return np.concatenate([x for x in [
[None]*(N // 2 + N % 2)*fill,
np.convolve(x, np.ones((N,))/N, mode='valid'),
[None]*(N // 2)*fill,
] if len(x)])评论区里的代码看起来很丑陋,我不想再发一条新答案了,因为已经有太多了,不过你可以把它复制粘贴到你的 IDE 里。 - Chaoste高效的解决方案
卷积比直接计算更好,但(我猜)它使用FFT,因此速度相对较慢。然而,特别适用于计算运行平均值的以下方法效果不错。
def running_mean(x, N):
cumsum = numpy.cumsum(numpy.insert(x, 0, 0))
return (cumsum[N:] - cumsum[:-N]) / float(N)
检查代码
In[3]: x = numpy.random.random(100000)
In[4]: N = 1000
In[5]: %timeit result1 = numpy.convolve(x, numpy.ones((N,))/N, mode='valid')
10 loops, best of 3: 41.4 ms per loop
In[6]: %timeit result2 = running_mean(x, N)
1000 loops, best of 3: 1.04 ms per loop
请注意,numpy.allclose(result1, result2) 的返回值为 True,表示两种方法是等价的。
警告:虽然使用 cumsum 方法速度更快,但会增加浮点误差,可能导致结果无效/不正确/不可接受。
# demonstrate loss of precision with only 100,000 points
np.random.seed(42)
x = np.random.randn(100000)+1e6
y1 = running_mean_convolve(x, 10)
y2 = running_mean_cumsum(x, 10)
assert np.allclose(y1, y2, rtol=1e-12, atol=0)
- 累积的点数越多,浮点误差就越大(所以1e5个点是可以察觉的,1e6个点更明显,超过1e6后可能需要重置累加器)。
- 你可以使用
np.longdouble来作弊,但对于较大数量的点数(大约>1e5,具体取决于数据),浮点误差仍然会变得显著。 - 您可以绘制误差曲线并观察它相对快速地增加。
- 卷积解决方案较慢,但不会出现这种浮点精度损失。
- uniform_filter1d解决方案比此cumsum解决方案更快,并且不会出现这种浮点精度损失。
13
running_mean([1,2,3], 2) 的结果为 array([1, 2])。将 x 替换为 [float(value) for value in x] 即可解决问题。 - ChrisWx包含浮点数,此解决方案的数值稳定性可能会成为问题。例如:running_mean(np.arange(int(1e7))[::-1] + 0.2, 1)[-1] - 0.2返回0.003125,而我们期望得到的是0.0。更多信息请参阅https://en.wikipedia.org/wiki/Loss_of_significance。 - Milan你可以使用 scipy.ndimage.uniform_filter1d:
import numpy as np
from scipy.ndimage import uniform_filter1d
N = 1000
x = np.random.random(100000)
y = uniform_filter1d(x, size=N)
uniform_filter1d:
- 输出与numpy数组具有相同的形状(即点数)
- 允许使用多种方式处理边界,其中
'reflect'是默认设置,但在我的情况下,我更喜欢使用'nearest'
该方法速度较快(几乎比np.convolve快50倍,比上述cumsum方法快2-5倍):
%timeit y1 = np.convolve(x, np.ones((N,))/N, mode='same')
100 loops, best of 3: 9.28 ms per loop
%timeit y2 = uniform_filter1d(x, size=N)
10000 loops, best of 3: 191 µs per loop
这里有3个函数,可以让您比较不同实现的错误/速度:
from __future__ import division
import numpy as np
import scipy.ndimage as ndi
def running_mean_convolve(x, N):
return np.convolve(x, np.ones(N) / float(N), 'valid')
def running_mean_cumsum(x, N):
cumsum = np.cumsum(np.insert(x, 0, 0))
return (cumsum[N:] - cumsum[:-N]) / float(N)
def running_mean_uniform_filter1d(x, N):
return ndi.uniform_filter1d(x, N, mode='constant', origin=-(N//2))[:-(N-1)]
3
uniform_filter1d、np.convolve和矩形以及np.cumsum后跟np.subtract进行了分析。我的结果是:(1)卷积最慢。(2)cumsum/subtract大约快20-30倍。(3)uniform_filter1d比cumsum/subtract快2-3倍。胜者绝对是uniform_filter1d。 - Trevor Boyd Smithuniform_filter1d 比 cumsum 解决方案更快(大约快2-5倍)。而且,与 cumsum 解决方案相比,uniform_filter1d 不会出现大量浮点误差。 (参考链接:https://dev59.com/g2Yr5IYBdhLWcg3wYZKD#BLOiEYcBWogLw_1bovvC) - Trevor Boyd Smith更新:下面的示例显示了旧版本的 pandas.rolling_mean 函数,该函数已在最新版本的 pandas 中被删除。现代等效的函数调用将使用 pandas.Series.rolling:
In [8]: pd.Series(x).rolling(window=N).mean().iloc[N-1:].values
Out[8]:
array([ 0.49815397, 0.49844183, 0.49840518, ..., 0.49488191,
0.49456679, 0.49427121])
Pandas比NumPy或SciPy更适合这个任务。它的函数rolling_mean可以方便地完成此任务,且当输入为数组时,它还会返回一个NumPy数组。
使用任何自定义的纯Python实现都很难在性能上超越rolling_mean。以下是性能示例与两种提议的解决方案进行比较:
In [1]: import numpy as np
In [2]: import pandas as pd
In [3]: def running_mean(x, N):
...: cumsum = np.cumsum(np.insert(x, 0, 0))
...: return (cumsum[N:] - cumsum[:-N]) / N
...:
In [4]: x = np.random.random(100000)
In [5]: N = 1000
In [6]: %timeit np.convolve(x, np.ones((N,))/N, mode='valid')
10 loops, best of 3: 172 ms per loop
In [7]: %timeit running_mean(x, N)
100 loops, best of 3: 6.72 ms per loop
In [8]: %timeit pd.rolling_mean(x, N)[N-1:]
100 loops, best of 3: 4.74 ms per loop
In [9]: np.allclose(pd.rolling_mean(x, N)[N-1:], running_mean(x, N))
Out[9]: True
还有一些不错的选项来处理边缘值。
6
df.rolling(windowsize).mean()。这个方法非常快速。对于一个有6,000行的数据系列,%timeit test1.rolling(20).mean() 返回 1000 loops, best of 3: 1.16 ms per loop. - Vloxdf.rolling() 目前已经能够正常运行,但问题是未来即使使用这种形式也将不支持 ndarrays。为了使用它,我们必须先将数据加载到 Pandas Dataframe 中。我希望看到这个函数被添加到 numpy 或 scipy.signal 中。 - Mike您可以使用以下公式来计算移动平均值:
import numpy as np
def runningMean(x, N):
y = np.zeros((len(x),))
for ctr in range(len(x)):
y[ctr] = np.sum(x[ctr:(ctr+N)])
return y/N
但这很慢。
幸运的是,numpy 包含一个 convolve 函数,我们可以用它来加速处理。运行均值等同于将 x 与一个长度为 N,所有元素都相等于 1/N 的向量进行卷积。Numpy 的 convolve 实现包括了起始瞬态,因此你必须移除前 N-1 个点:
def runningMeanFast(x, N):
return np.convolve(x, np.ones((N,))/N)[(N-1):]
在我的电脑上,快速版本比慢速版本快20-30倍,具体取决于输入向量的长度和平均窗口的大小。
请注意,convolve函数确实包括一个'same'模式,它似乎可以解决起始瞬态问题,但它将其分为开头和结尾两部分。
5
convolve中使用mode ='valid',它不需要任何后处理。 - lapismode='valid'会从两端移除瞬态,对吗?如果len(x)=10且N=4,对于运行平均值,我想要10个结果,但是valid只返回7个。 - mtrwmodes = ('full', 'same', 'valid'); [plot(convolve(ones((200,)), ones((50,))/50, mode=m)) for m in modes]; axis([-10, 251, -.1, 1.1]); legend(modes, loc='lower center')(导入pyplot和numpy)。 - lapisrunningMean 当你在数组的右侧使用 x[ctr:(ctr+N)] 时,是否会出现用零进行平均的副作用。 - mrgloomrunningMeanFast 也存在这个边界效应问题。 - mrgloom以下代码是一种快速简洁的解决方案,只需要一个循环即可完成整个过程,而且不需要任何依赖。
mylist = [1, 2, 3, 4, 5, 6, 7]
N = 3
cumsum, moving_aves = [0], []
for i, x in enumerate(mylist, 1):
cumsum.append(cumsum[i-1] + x)
if i>=N:
moving_ave = (cumsum[i] - cumsum[i-N])/N
#can do stuff with moving_ave here
moving_aves.append(moving_ave)
3
一个用于计算的Python模块
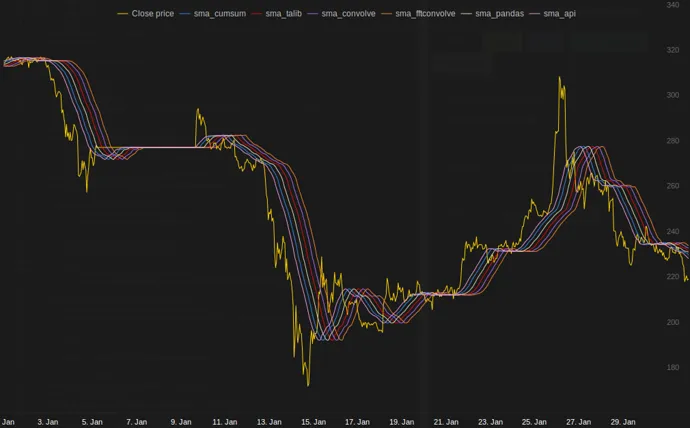
在Tradewave.net的测试中,TA-lib总是获胜:
import talib as ta
import numpy as np
import pandas as pd
import scipy
from scipy import signal
import time as t
PAIR = info.primary_pair
PERIOD = 30
def initialize():
storage.reset()
storage.elapsed = storage.get('elapsed', [0,0,0,0,0,0])
def cumsum_sma(array, period):
ret = np.cumsum(array, dtype=float)
ret[period:] = ret[period:] - ret[:-period]
return ret[period - 1:] / period
def pandas_sma(array, period):
return pd.rolling_mean(array, period)
def api_sma(array, period):
# this method is native to Tradewave and does NOT return an array
return (data[PAIR].ma(PERIOD))
def talib_sma(array, period):
return ta.MA(array, period)
def convolve_sma(array, period):
return np.convolve(array, np.ones((period,))/period, mode='valid')
def fftconvolve_sma(array, period):
return scipy.signal.fftconvolve(
array, np.ones((period,))/period, mode='valid')
def tick():
close = data[PAIR].warmup_period('close')
t1 = t.time()
sma_api = api_sma(close, PERIOD)
t2 = t.time()
sma_cumsum = cumsum_sma(close, PERIOD)
t3 = t.time()
sma_pandas = pandas_sma(close, PERIOD)
t4 = t.time()
sma_talib = talib_sma(close, PERIOD)
t5 = t.time()
sma_convolve = convolve_sma(close, PERIOD)
t6 = t.time()
sma_fftconvolve = fftconvolve_sma(close, PERIOD)
t7 = t.time()
storage.elapsed[-1] = storage.elapsed[-1] + t2-t1
storage.elapsed[-2] = storage.elapsed[-2] + t3-t2
storage.elapsed[-3] = storage.elapsed[-3] + t4-t3
storage.elapsed[-4] = storage.elapsed[-4] + t5-t4
storage.elapsed[-5] = storage.elapsed[-5] + t6-t5
storage.elapsed[-6] = storage.elapsed[-6] + t7-t6
plot('sma_api', sma_api)
plot('sma_cumsum', sma_cumsum[-5])
plot('sma_pandas', sma_pandas[-10])
plot('sma_talib', sma_talib[-15])
plot('sma_convolve', sma_convolve[-20])
plot('sma_fftconvolve', sma_fftconvolve[-25])
def stop():
log('ticks....: %s' % info.max_ticks)
log('api......: %.5f' % storage.elapsed[-1])
log('cumsum...: %.5f' % storage.elapsed[-2])
log('pandas...: %.5f' % storage.elapsed[-3])
log('talib....: %.5f' % storage.elapsed[-4])
log('convolve.: %.5f' % storage.elapsed[-5])
log('fft......: %.5f' % storage.elapsed[-6])
结果:
[2015-01-31 23:00:00] ticks....: 744
[2015-01-31 23:00:00] api......: 0.16445
[2015-01-31 23:00:00] cumsum...: 0.03189
[2015-01-31 23:00:00] pandas...: 0.03677
[2015-01-31 23:00:00] talib....: 0.00700 # <<< Winner!
[2015-01-31 23:00:00] convolve.: 0.04871
[2015-01-31 23:00:00] fft......: 0.22306
4
如需立即可用的解决方案,请参见https://scipy-cookbook.readthedocs.io/items/SignalSmooth.html。
该方案提供了使用flat窗口类型的移动平均值。请注意,这比简单的自行卷积方法要复杂一些,因为它试图通过反射来处理数据开头和结尾的问题(在您的情况下可能有用,也可能无用...)。
首先,您可以尝试:
a = np.random.random(100)
plt.plot(a)
b = smooth(a, window='flat')
plt.plot(b)
4
numpy.convolve,唯一的区别在于改变序列。 - Alleoy=np.convolve(w/w.sum(),s,mode='same');
return y[window_len-1:-(window_len-1)] - Christian O'Reillyw是窗口大小,s是数据吗? - Demis我知道这是一个旧问题,但这里有一个解决方案,它不使用任何额外的数据结构或库。它在输入列表的元素数量上是线性的,我无法想到其他更有效的方法(实际上,如果有人知道一种更好的分配结果的方法,请告诉我)。
注意:使用numpy数组而不是列表会快得多,但我想消除所有依赖性。也可以通过多线程执行来提高性能。
该函数假设输入列表是一维的,所以请小心。
### Running mean/Moving average
def running_mean(l, N):
sum = 0
result = list( 0 for x in l)
for i in range( 0, N ):
sum = sum + l[i]
result[i] = sum / (i+1)
for i in range( N, len(l) ):
sum = sum - l[i-N] + l[i]
result[i] = sum / N
return result
示例
假设我们有一个列表data = [1,2,3,4,5,6],我们想要对其进行周期为3的滚动平均值计算,并且您还希望输出列表与输入列表大小相同(这通常是最常见的情况)。
第一个元素的索引为0,因此滚动平均应该计算索引为-2、-1和0的元素。显然,我们没有data[-2]和data[-1](除非您想使用特殊的边界条件),因此我们假设这些元素为0。这相当于对列表进行零填充,除了我们实际上不填充它,只是跟踪需要填充的索引(从0到N-1)。
因此,对于前N个元素,我们只需将元素累加到累加器中。
result[0] = (0 + 0 + 1) / 3 = 0.333 == (sum + 1) / 3
result[1] = (0 + 1 + 2) / 3 = 1 == (sum + 2) / 3
result[2] = (1 + 2 + 3) / 3 = 2 == (sum + 3) / 3
从第N+1个元素开始,简单的累加方法不再适用。我们期望result[3]=(2+3+4)/3=3,但这与(sum+4)/3=3.333不同。
计算正确值的方法是从sum+4中减去data[0]=1,因此得到sum+4-1=9。
这是因为目前sum=data[0]+data[1]+data[2],但对于每个i >= N也是如此,因为在减法之前,sum=data[i-N]+...+data[i-2]+data[i-1]。
我认为可以通过使用bottleneck优雅地解决这个问题。
以下是基本示例:
import numpy as np
import bottleneck as bn
a = np.random.randint(4, 1000, size=100)
mm = bn.move_mean(a, window=5, min_count=1)
"mm" 是 "a" 的移动平均值。
"window" 是考虑移动平均的最大条目数。
"min_count" 是考虑移动平均的最小条目数(例如,对于前几个元素或如果数组具有nan值)。
好的部分是Bottleneck帮助处理nan值并且也非常高效。
1
原文链接