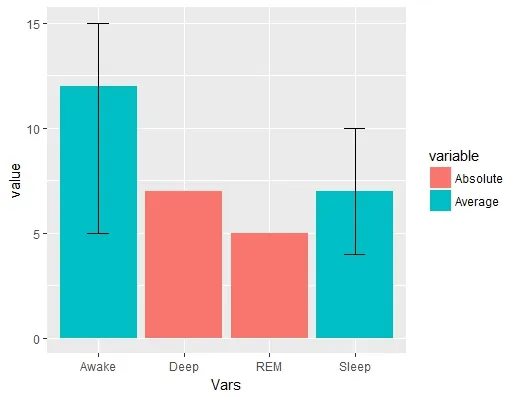
- 旋转CSV数据中的行和列以获得线程A very simple histogram with R?所需的相应数据结构(高表)并使用
ggplot绘制。 绘制事件的直方图作为
Absolute变量XOR(Average,Min,Max)- 如果仅有绝对值,则只需在直方图中绘制绝对值。
- 如果是(平均值、最小值和最大值),则在直方图中绘制它们与须(=whisker plot), 其中须的限制由最小值和最大值确定。
数据
initially,
data.csv"Vars" , "Sleep", "Awake", "REM", "Deep" "Absolute", , , 5 , 7 "Average" , 7 , 12 , , "Min" , 4 , 5 , , "Max" , 10 , 15 , ,data after reshaping visually
V1 V2 V3 V4 Vars Absolute Average Min Max Sleep <NA> 7 4 10 Awake <NA> 12 5 15 REM 5 <NA> <NA> <NA> Deep 7 <NA> <NA> <NA>data after reshaping for R
data <- structure(list(V1 = structure(c(3L, NA, NA, 1L, 2L), .Names = c("Vars", "Sleep", "Awake", "REM", "Deep"), .Label = c(" 5", " 7", "Absolute" ), class = "factor"), V2 = structure(c(3L, 2L, 1L, NA, NA), .Names = c("Vars", "Sleep", "Awake", "REM", "Deep"), .Label = c("12", " 7", "Average " ), class = "factor"), V3 = structure(c(3L, 1L, 2L, NA, NA), .Names = c("Vars", "Sleep", "Awake", "REM", "Deep"), .Label = c(" 4", " 5", "Min " ), class = "factor"), V4 = structure(c(3L, 1L, 2L, NA, NA), .Names = c("Vars", "Sleep", "Awake", "REM", "Deep"), .Label = c("10", "15", "Max " ), class = "factor")), .Names = c("V1", "V2", "V3", "V4"), row.names = c("Vars", "Sleep", "Awake", "REM", "Deep"), class = "data.frame")
带有调试代码的R代码
dat.m <- read.csv("data.csv")
# rotate rows and columns
dat.m <- as.data.frame(t(dat.m)) # https://dev59.com/jGs05IYBdhLWcg3wQvyq#7342329 Comment 42-
library("reshape2")
dat.m <- melt(dat.m, id.vars="Vars")
## Just plot values existing there correspondingly
library("ggplot2")
# https://stackoverflow.com/a/25584792/54964
# TODO following
#ggplot(dat.m, aes(x = "Vars", y = value,fill=variable))
错误
Error: id variables not found in data: Vars
Execution halted
R: 3.3.3,3.4.0(后移版本)
操作系统:Debian 8.7
使用sessionInfo()在加载这两个包后,重塑R reshape2,ggplot2等。
Platform: x86_64-pc-linux-gnu (64-bit)
locale:
[1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C
[3] LC_TIME=en_US.UTF-8 LC_COLLATE=en_US.UTF-8
[5] LC_MONETARY=en_US.UTF-8 LC_MESSAGES=en_US.UTF-8
[7] LC_PAPER=en_US.UTF-8 LC_NAME=C
[9] LC_ADDRESS=C LC_TELEPHONE=C
[11] LC_MEASUREMENT=en_US.UTF-8 LC_IDENTIFICATION=C
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] ggplot2_2.1.0 reshape2_1.4.2
loaded via a namespace (and not attached):
[1] colorspace_1.3-2 scales_0.4.1 magrittr_1.5 plyr_1.8.4
[5] tools_3.3.3 gtable_0.2.0 Rcpp_0.12.10 stringi_1.1.5
[9] grid_3.3.3 stringr_1.2.0 munsell_0.4.3
测试HaberdashPI的提案
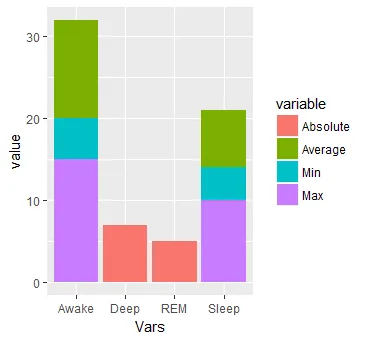
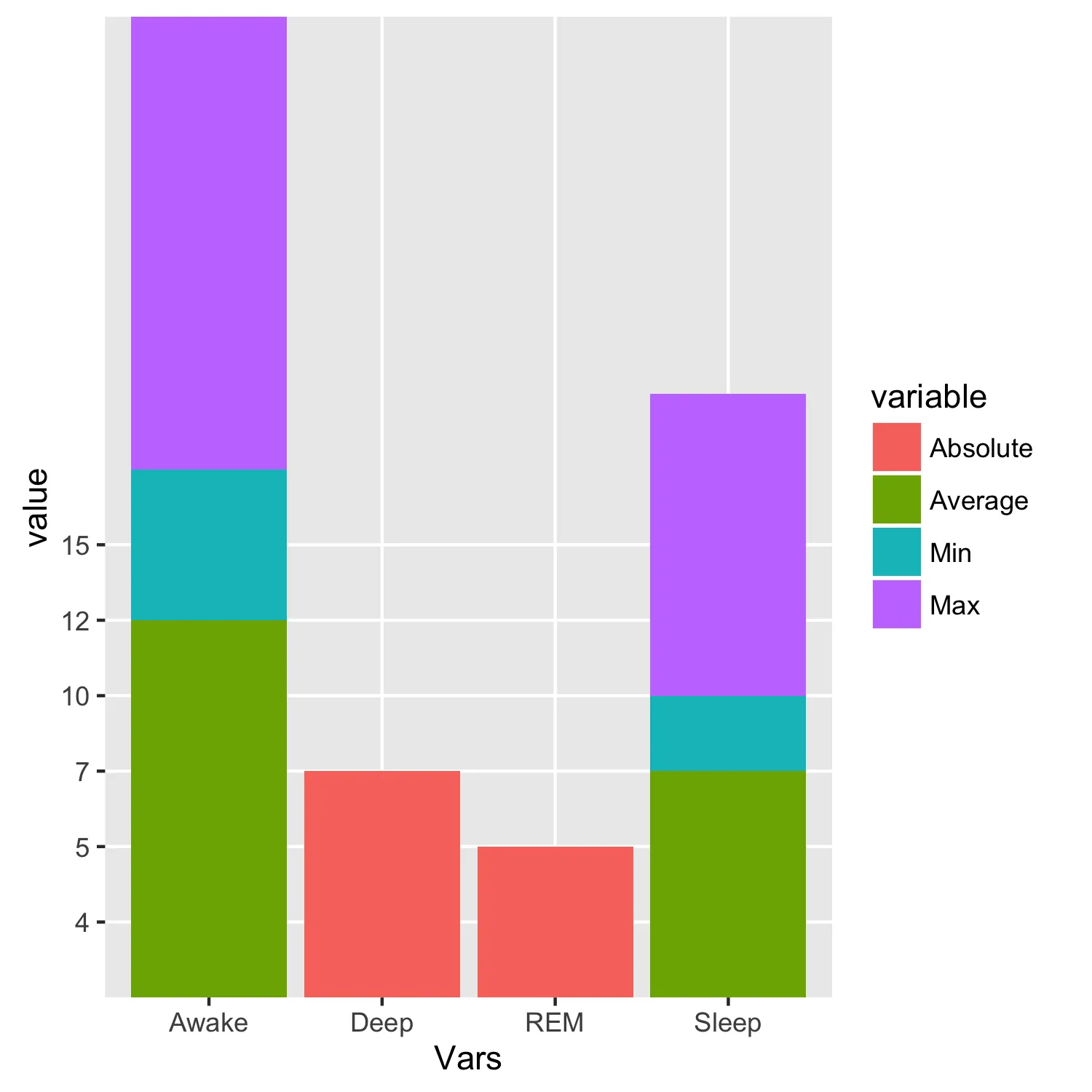
图1中的输出中,Sleep和Awake中的绝对值错误。
如果是NA,则将值设置为零。
图1 HaberdashPI的提案输出不如预期。
dat.m在转置之前的数据结构
'data.frame': 4 obs. of 5 variables:
$ Absolute: Factor w/ 2 levels " 5"," 7": NA NA 1 2
..- attr(*, "names")= chr "Sleep" "Awake" "REM" "Deep"
$ Average : Factor w/ 2 levels "12"," 7": 2 1 NA NA
..- attr(*, "names")= chr "Sleep" "Awake" "REM" "Deep"
$ Min : Factor w/ 2 levels " 4"," 5": 1 2 NA NA
..- attr(*, "names")= chr "Sleep" "Awake" "REM" "Deep"
$ Max : Factor w/ 2 levels "10","15": 1 2 NA NA
..- attr(*, "names")= chr "Sleep" "Awake" "REM" "Deep"
$ Vars : chr "Sleep" "Awake" "REM" "Deep"
Absolute Average Min Max Vars
Sleep <NA> 7 4 10 Sleep
Awake <NA> 12 5 15 Awake
REM 5 <NA> <NA> <NA> REM
Deep 7 <NA> <NA> <NA> Deep
转置后dat.m的数据结构
'data.frame': 16 obs. of 3 variables:
$ Vars : chr "Sleep" "Awake" "REM" "Deep" ...
$ variable: Factor w/ 4 levels "Absolute","Average ",..: 1 1 1 1 2 2 2 2 3 3 ...
$ value : chr NA NA " 5" " 7" ...
Vars variable value
1 Sleep Absolute <NA>
2 Awake Absolute <NA>
3 REM Absolute 5
4 Deep Absolute 7
5 Sleep Average 7
6 Awake Average 12
7 REM Average <NA>
8 Deep Average <NA>
9 Sleep Min 4
10 Awake Min 5
11 REM Min <NA>
12 Deep Min <NA>
13 Sleep Max 10
14 Awake Max 15
15 REM Max <NA>
16 Deep Max <NA>
测试 akash87 的 提案
代码
ds <- dat.m
str(ds)
ds
ds$variable
ds$variable %in% c("Min","Max")
错误输出是因为最后所有的
False。 $ Vars : chr "Sleep" "Awake" "REM" "Deep" ...
$ variable: Factor w/ 4 levels "Absolute","Average ",..: 1 1 1 1 2 2 2 2 3 3 ...
$ value : chr NA NA " 5" " 7" ...
Vars variable value
1 Sleep Absolute <NA>
2 Awake Absolute <NA>
3 REM Absolute 5
4 Deep Absolute 7
5 Sleep Average 7
6 Awake Average 12
7 REM Average <NA>
8 Deep Average <NA>
9 Sleep Min 4
10 Awake Min 5
11 REM Min <NA>
12 Deep Min <NA>
13 Sleep Max 10
14 Awake Max 15
15 REM Max <NA>
16 Deep Max <NA>
[1] "hello 3"
[1] Absolute Absolute Absolute Absolute Average Average Average Average
[9] Min Min Min Min Max Max Max Max
Levels: Absolute Average Min Max
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE
因为存在错误传递,所以执行 ds[ds$variable %in% c("Min","Max"), ] 将会得到 False 的输出。
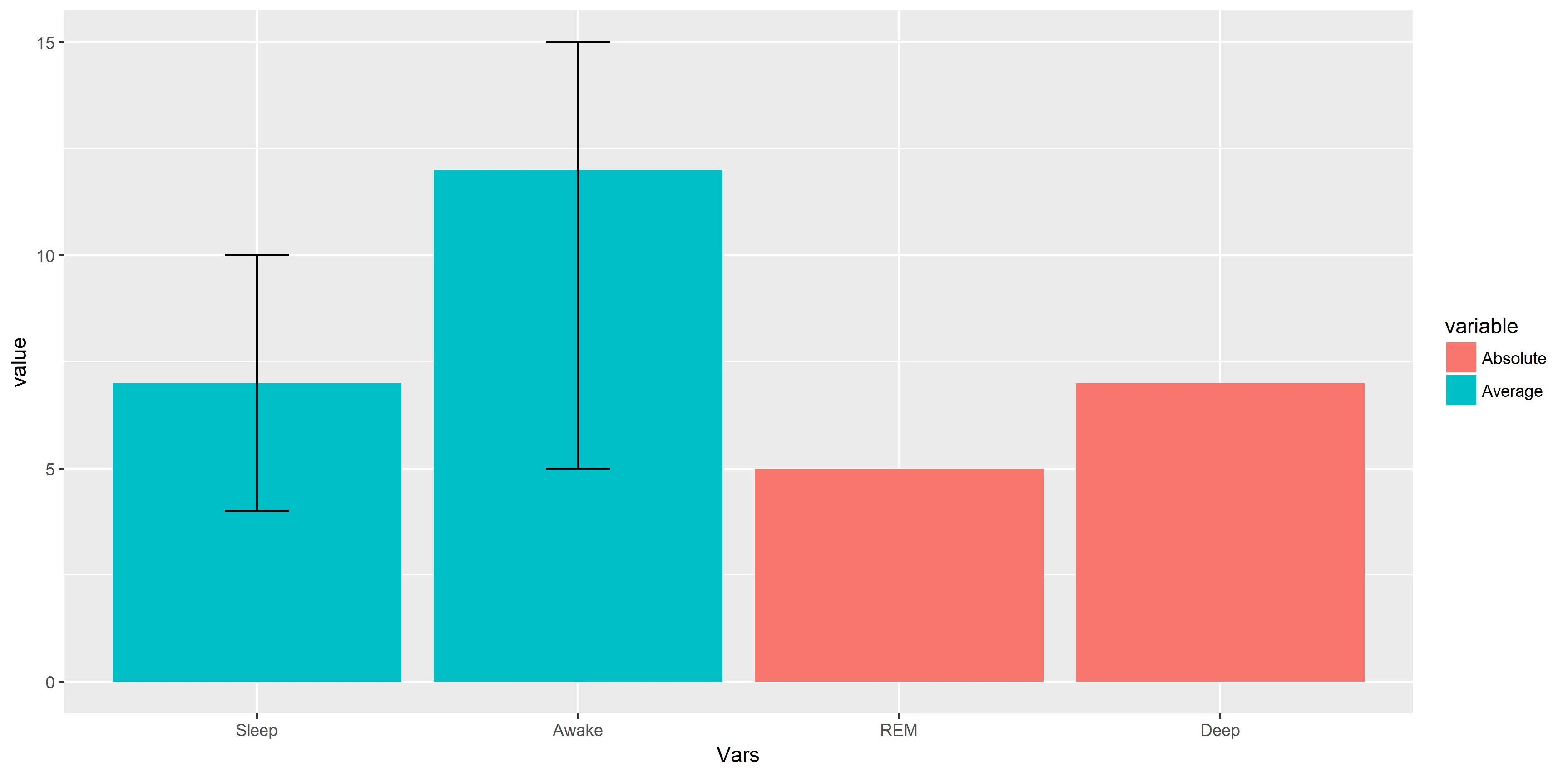
测试Uwe的提案
使用明确的 data.table::dcast 代码和两次 data.table::melt。在 molten <- ... 之前打印出 sessionInfo()。请注意,因为错误来自于 molten <- ... 这一行,所以尚未加载 library(ggplot2)。
$ Rscript test111.r
Vars "Average" "Max" "Min" Absolute
1: Sleep 7 10 4 NA
2: Awake 12 15 5 NA
3: REM NA NA NA 5
4: Deep NA NA NA 7
R version 3.4.0 (2017-04-21)
Platform: x86_64-pc-linux-gnu (64-bit)
Running under: Debian GNU/Linux 8 (jessie)
Matrix products: default
BLAS: /usr/lib/openblas-base/libblas.so.3
LAPACK: /usr/lib/libopenblasp-r0.2.12.so
locale:
[1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C
[3] LC_TIME=en_US.UTF-8 LC_COLLATE=en_US.UTF-8
[5] LC_MONETARY=en_US.UTF-8 LC_MESSAGES=en_US.UTF-8
[7] LC_PAPER=en_US.UTF-8 LC_NAME=C
[9] LC_ADDRESS=C LC_TELEPHONE=C
[11] LC_MEASUREMENT=en_US.UTF-8 LC_IDENTIFICATION=C
attached base packages:
[1] stats graphics grDevices utils datasets base
other attached packages:
[1] data.table_1.10.4
loaded via a namespace (and not attached):
[1] compiler_3.4.0 methods_3.4.0
Error in melt.data.table(transposed, measure.vars = c("Absolute", "Average")) :
One or more values in 'measure.vars' is invalid.
Calls: <Anonymous> -> melt.data.table
Execution halted
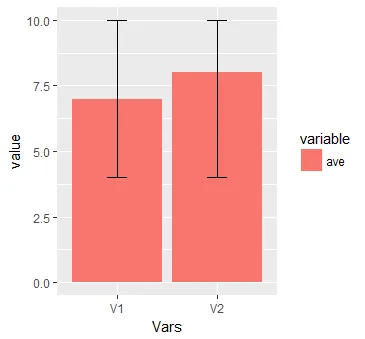
使用测试代码2测试Uwe的建议
代码
molten <- structure(list(Vars = structure(c(1L, 2L, 1L, 2L, 1L, 2L), class = "factor", .Label = c("V1", "V2")), variable = structure(c(1L, 1L, 2L, 2L, 3L, 3L), class = "factor", .Label = c("ave", "ave_max", "lepo")), value = c(7L, 8L, 10L, 10L, 4L, 4L)), .Names = c("Vars", "variable", "value"), row.names = c(NA, -6L), class = c("data.table", "data.frame"))
print(molten)
library(ggplot2)
ggplot(molten, aes(x = Vars, y = value, fill = variable, ymin = lepo, ymax = ave_max)) +
geom_col() + geom_errorbar(width = 0.2)
输出
Vars variable value
1 V1 ave 7
2 V2 ave 8
3 V1 ave_max 10
4 V2 ave_max 10
5 V1 lepo 4
6 V2 lepo 4
Error in FUN(X[[i]], ...) : object 'lepo' not found
Calls: <Anonymous> ... by_layer -> f -> <Anonymous> -> f -> lapply -> FUN -> FUN
Execution halted