我正在尝试使用ecdf()函数绘制一个CDF图,以下是代码:
> x<-ecdf(data$V6)
> summary(x)
Empirical CDF: 2402 unique values with summary
Min. 1st Qu. Median Mean 3rd Qu. Max.
3392 71870 120100 386100 219000 158600000
plot(x, log='x')
Error in plot.window(...) : Logarithmic axis must have positive limits
我的数据集呈指数增长,因此我希望在x轴上使用对数刻度。当我不使用 log="x" 时,它可以工作,但绘图效果不好。我需要x轴是对数的,有什么办法吗?
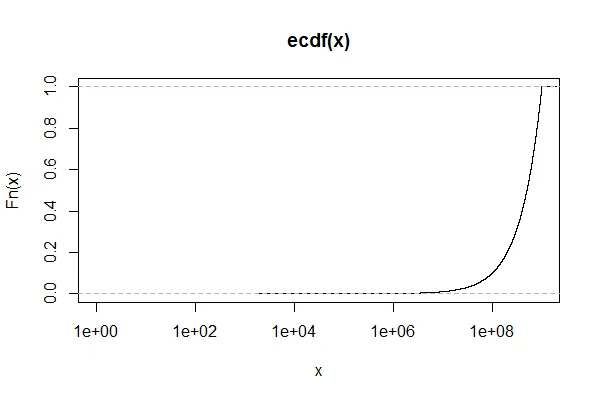
xlim=c(0,158600000)但是出现了另一个错误:nonfinite axis limits [GScale(-inf,8.2003,1, .); log=1]。 - Nasir