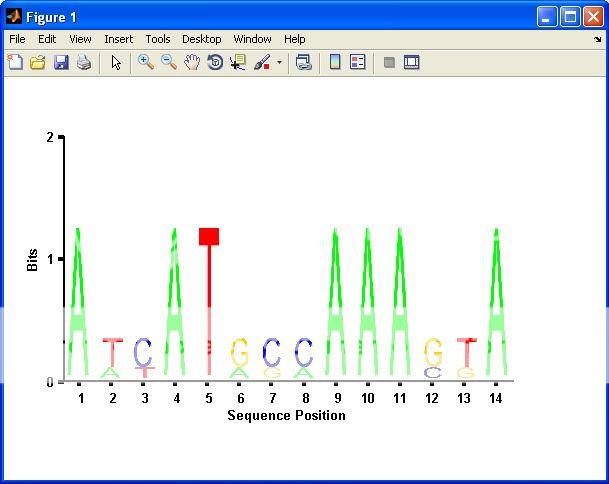
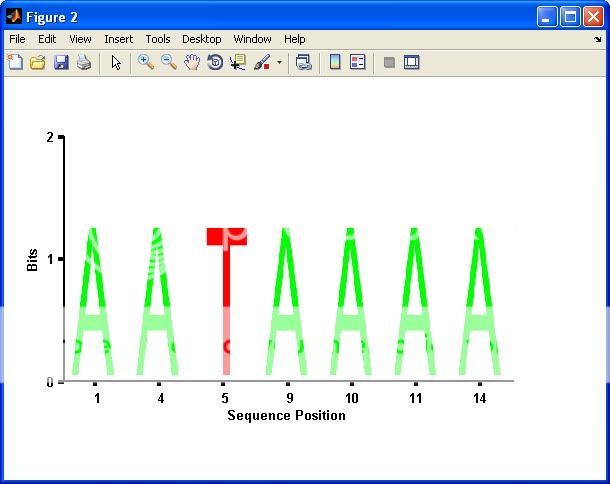
所以我创建了另一种解决方案,同时采用了yuk和gnovice的方法。当我试着使用这个解决方案时,我意识到我真的很想能够将输出用作"子图",并且可以任意更改字母的颜色。
由于yuk使用编程方式放置的带有嵌入式字母的轴对象,如果要修改他的代码以绘制到任意轴对象中,这将非常麻烦(虽然不是不可能)。由于gnovice的解决方案从预先创建的文件中读取字母,所以难以修改代码以对任意颜色方案或字母选择运行。因此,我的解决方案使用了yuk解决方案中的"字母生成"代码和gnovice解决方案中的"图像叠加"方法。
还有大量的参数解析和检查。下面是我的综合解决方案… 我只包括它是为了完整性,显然我不能赢得我的奖励。我将让社区决定奖励,并在时间限制结束时将赏金颁给评分最高的人... 如果打成平局,我会把它给予声誉最低的人(他们可能更需要它)。
function [npos, handle] = SeqLogoFig(SEQ, varargin)
ALPHA = 'nt';
MAX_BITS = 2.5;
RES = [200 80];
CUTOFF = [];
TOPN = [];
rm_inds = [];
colors = [];
handle = [];
npos = [];
for i = 1:2:length(varargin)
if strcmpi(varargin{i}, 'alphabet')
ALPHA = varargin{i+1};
elseif strcmpi(varargin{i}, 'cutoff')
CUTOFF = varargin{i+1};
rm_inds = [rm_inds i, i+1];
elseif strcmpi(varargin{i}, 'colors')
colors = varargin{i+1};
rm_inds = [rm_inds i, i+1];
elseif strcmpi(varargin{i}, 'axes_handle')
handle = varargin{i+1};
rm_inds = [rm_inds i, i+1];
elseif strcmpi(varargin{i}, 'top-n')
TOPN = varargin{i+1};
rm_inds = [rm_inds i, i+1];
elseif strcmpi(varargin{i}, 'inds')
npos = varargin{i+1};
rm_inds = [rm_inds i, i+1];
end
end
if ~isempty(rm_inds)
varargin(rm_inds) = [];
end
if isempty(colors)
colors = GetColors(ALPHA);
end
if strcmpi(ALPHA, 'nt')
MAX_BITS = 2.5;
elseif strcmpi(ALPHA, 'aa')
MAX_BITS = 4.5;
end
if isempty(CUTOFF)
CUTOFF = 0.5*MAX_BITS;
end
wm = seqlogo(SEQ, varargin{:}, 'displaylogo', false);
letters = wm{1};
letter_wins = cell(size(letters));
[~, loc] = ismember(letters, colors(:,1));
loc(loc == 0) = size(colors,1);
clr = cell2mat(colors(loc, 2));
for t = 1:numel(letters)
hf = figure('position',[200 200 100 110],'color','w');
ha = axes('parent',hf, 'visible','off','position',[0 0 1 1]);
ht = text(50,55,letters(t),'color',clr(t,:),'units','pixels',...
'fontsize',100,'fontweight','norm',...
'vertical','mid','horizontal','center');
F = getframe(hf);
img = F.cdata;
m = any(img < 255,3);
m(any(m,2),any(m,1))=1;
letter_wins{t} = reshape(img(repmat(m,[1 1 3])),[sum(any(m,2)) sum(any(m,1)) 3]);
close(hf);
end
wmat = wm{2};
if isempty(npos)
if isempty(TOPN)
npos = find(any(wmat>CUTOFF,1));
else
[~, i] = sort(max(wmat,[],1), 'descend');
npos = sort(i(1:TOPN));
end
end
fig_data = 255*ones(RES(1), RES(2)*(length(npos)+1)+length(npos)*2,3);
bitscores = linspace(0, MAX_BITS, size(fig_data,1));
tick_pos = zeros(length(npos),1);
for i=1:length(npos)
[wms idx] = sort(wmat(:,npos(i)), 'descend');
bits = [flipud(cumsum(flipud(wms))); 0];
let_data = letter_wins(idx(wms>0));
for s=1:length(let_data)
start_pos = find(bitscores>=bits(s),1);
end_pos = find(bitscores<=bits(s+1),1, 'last');
if isempty(start_pos) || isempty(end_pos) || end_pos > start_pos
continue
end
img_win = imresize(let_data{s}, [start_pos-end_pos, RES(2)]);
fig_data(start_pos-1:-1:end_pos, (i*RES(2)-RES(2)*.5:i*RES(2)+RES(2)*.5-1)+2*i,:) = img_win;
end
tick_pos(i) = i*RES(2)+2*i;
end
if ~isempty(handle)
image(handle,[0 size(fig_data,2)], [0 MAX_BITS],fig_data./255)
else
handle = image([0 size(fig_data,2)], [0 MAX_BITS],fig_data./255);
end
set(gca, 'ydir', 'normal', 'xtick', tick_pos, ...
'userdata', tick_pos, 'xticklabel', npos);
xlabel('position')
ylabel('bits')
function colors = GetColors(alpha)
if strcmpi(alpha, 'nt')
colors = cell(6,2);
colors(1,:) = {'A', [0 1 0]};
colors(2,:) = {'C', [0 0 1]};
colors(3,:) = {'G', [1 1 0]};
colors(4,:) = {'T', [1 0 0]};
colors(5,:) = {'U', [1 0 0]};
colors(6,:) = {'', [1 0 1]};
elseif strcmpi(alpha, 'aa')
colors = cell(21,2);
colors(1,:) = {'G', [0 1 0]};
colors(2,:) = {'S', [0 1 0]};
colors(3,:) = {'T', [0 1 0]};
colors(4,:) = {'Y', [0 1 0]};
colors(5,:) = {'C', [0 1 0]};
colors(6,:) = {'Q', [0 1 0]};
colors(7,:) = {'N', [0 1 0]};
colors(8,:) = {'A', [1 165/255 0]};
colors(9,:) = {'V', [1 165/255 0]};
colors(10,:) = {'L', [1 165/255 0]};
colors(11,:) = {'I', [1 165/255 0]};
colors(12,:) = {'P', [1 165/255 0]};
colors(13,:) = {'W', [1 165/255 0]};
colors(14,:) = {'F', [1 165/255 0]};
colors(15,:) = {'M', [1 165/255 0]};
colors(16,:) = {'D', [1 0 0]};
colors(17,:) = {'E', [1 0 0]};
colors(18,:) = {'K', [0 0 1]};
colors(19,:) = {'R', [0 0 1]};
colors(20,:) = {'H', [0 0 1]};
colors(21,:) = {'', [210/255 180/255 140/255]};
else
error('SeqLogoFigure:BADALPHA', ...
'An unknown alphabet was provided: %s', alpha)
end
我已将此提交给Mathworks FileExchange,一旦获得批准,我会发布链接。
唯一令我困扰的是,在创建字母图像时,它会以快速的速度显示小窗口。如果有人知道可以避免这种情况的技巧,我很想听听。
编辑:Mathworks已经批准了我的提交文件...您可以在FileExchange这里下载:
http://www.mathworks.com/matlabcentral/fileexchange/27124