关于绘制置信区间有很多答案。
我正在阅读Lourme A. et al (2016)的论文,我想像该论文中的图2一样绘制90%置信边界和10%异常点: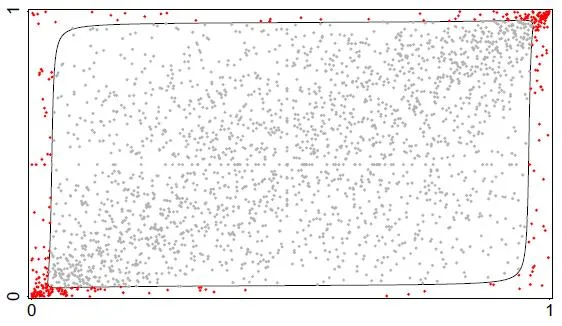 。
。
library("MASS")
library(copula)
set.seed(612)
n <- 1000 # length of sample
d <- 2 # dimension
# random vector with uniform margins on (0,1)
u1 <- runif(n, min = 0, max = 1)
u2 <- runif(n, min = 0, max = 1)
u = matrix(c(u1, u2), ncol=d)
Rg <- cor(u) # d-by-d correlation matrix
Rg1 <- ginv(Rg) # inv. matrix
# round(Rg %*% Rg1, 8) # check
# the multivariate c.d.f of u is a Gaussian copula
# with parameter Rg[1,2]=0.02876654
normal.cop = normalCopula(Rg[1,2], dim=d)
fit.cop = fitCopula(normal.cop, u, method="itau") #fitting
# Rg.hat = fit.cop@estimate[1]
# [1] 0.03097071
sim = rCopula(n, normal.cop) # in (0,1)
# Taking the quantile function of N1(0, 1)
y1 <- qnorm(sim[,1], mean = 0, sd = 1)
y2 <- qnorm(sim[,2], mean = 0, sd = 1)
par(mfrow=c(2,2))
plot(y1, y2, col="red"); abline(v=mean(y1), h=mean(y2))
plot(sim[,1], sim[,2], col="blue")
hist(y1); hist(y2)
参考文献。Lourme, A.,F. Maurer(2016)在风险管理框架中测试高斯和学生t簇的方法。经济建模。
问题。 有人可以帮助我解释方程中变量v =(v_1,...,v_d)和G(v_1),...,G(v_d)吗?
我认为v是非随机矩阵,其尺寸应为$k^2$(网格点)乘以d = 2(维度)。例如,
axis_x <- seq(0, 1, 0.1) # 11 grid points
axis_y <- seq(0, 1, 0.1) # 11 grid points
v <- expand.grid(axis_x, axis_y)
plot(v, type = "p")