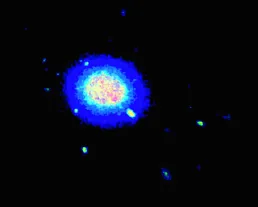
感谢mwaskon推荐mayavi库。
我按如下方式在mayavi中重新创建了密度散点图:
import numpy as np
from scipy import stats
from mayavi import mlab
mu, sigma = 0, 0.1
x = 10*np.random.normal(mu, sigma, 5000)
y = 10*np.random.normal(mu, sigma, 5000)
z = 10*np.random.normal(mu, sigma, 5000)
xyz = np.vstack([x,y,z])
kde = stats.gaussian_kde(xyz)
density = kde(xyz)
figure = mlab.figure('DensityPlot')
pts = mlab.points3d(x, y, z, density, scale_mode='none', scale_factor=0.07)
mlab.axes()
mlab.show()
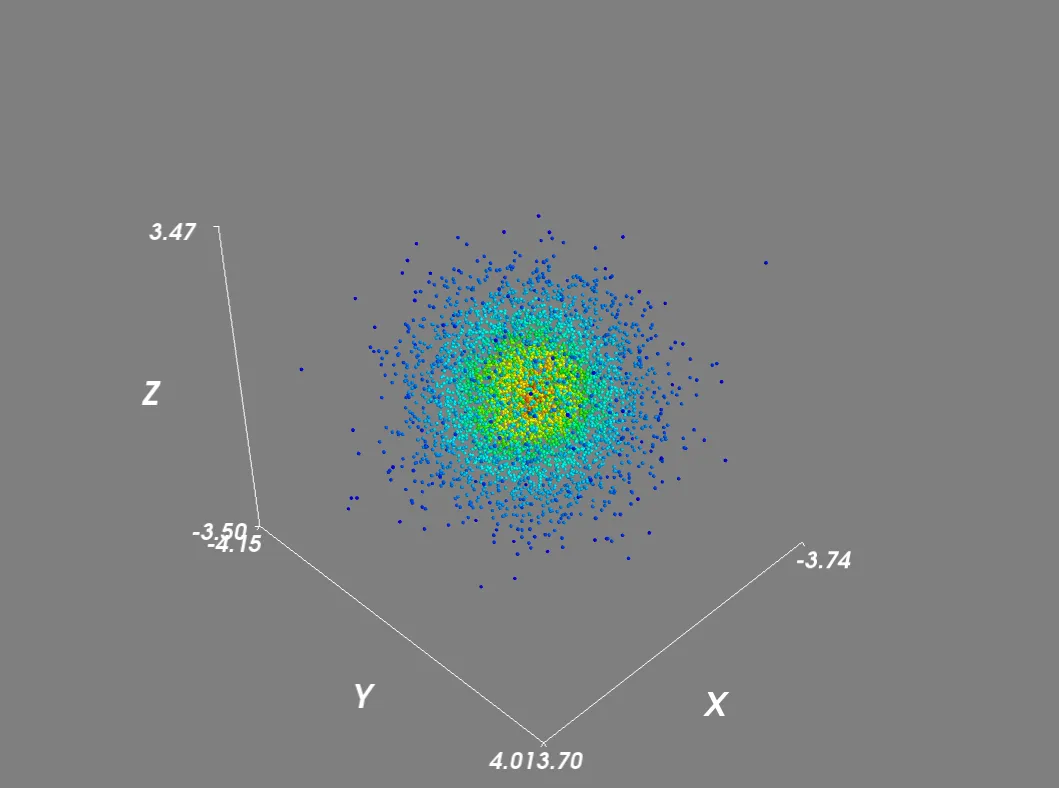
将scale_mode设置为“none”可以防止图形按照密度向量成比例缩放。此外,对于大型数据集,我禁用了场景渲染,并使用掩码减少了点的数量。
figure = mlab.figure('DensityPlot')
figure.scene.disable_render = True
pts = mlab.points3d(x, y, z, density, scale_mode='none', scale_factor=0.07)
mask = pts.glyph.mask_points
mask.maximum_number_of_points = x.size
mask.on_ratio = 1
pts.glyph.mask_input_points = True
figure.scene.disable_render = False
mlab.axes()
mlab.show()
接下来,要在网格上评估高斯核密度估计:
import numpy as np
from scipy import stats
from mayavi import mlab
mu, sigma = 0, 0.1
x = 10*np.random.normal(mu, sigma, 5000)
y = 10*np.random.normal(mu, sigma, 5000)
z = 10*np.random.normal(mu, sigma, 5000)
xyz = np.vstack([x,y,z])
kde = stats.gaussian_kde(xyz)
xmin, ymin, zmin = x.min(), y.min(), z.min()
xmax, ymax, zmax = x.max(), y.max(), z.max()
xi, yi, zi = np.mgrid[xmin:xmax:30j, ymin:ymax:30j, zmin:zmax:30j]
coords = np.vstack([item.ravel() for item in [xi, yi, zi]])
density = kde(coords).reshape(xi.shape)
figure = mlab.figure('DensityPlot')
grid = mlab.pipeline.scalar_field(xi, yi, zi, density)
min = density.min()
max=density.max()
mlab.pipeline.volume(grid, vmin=min, vmax=min + .5*(max-min))
mlab.axes()
mlab.show()
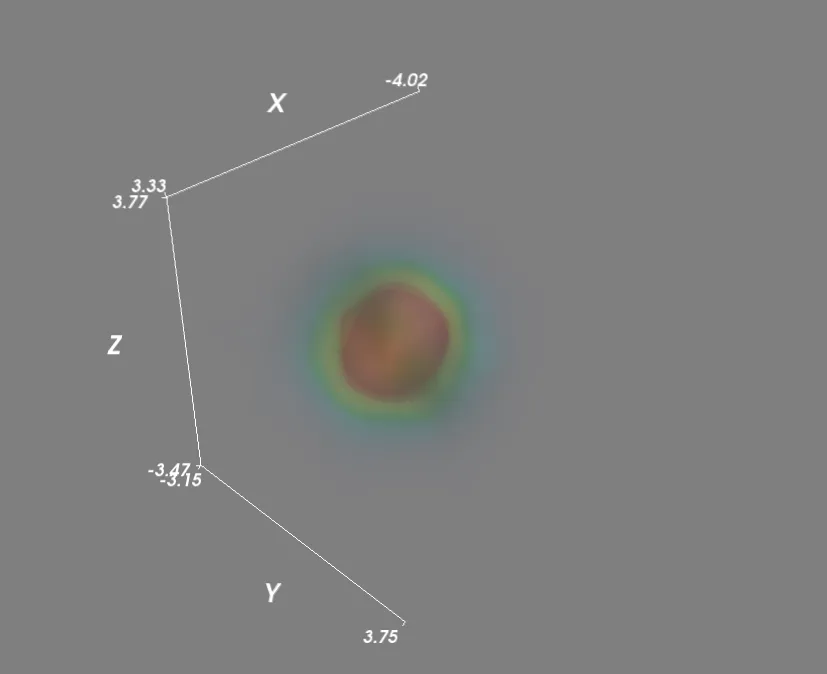
为了最终改进,我通过并行调用kde函数加快了核密度函数的评估。
import numpy as np
from scipy import stats
from mayavi import mlab
import multiprocessing
def calc_kde(data):
return kde(data.T)
mu, sigma = 0, 0.1
x = 10*np.random.normal(mu, sigma, 5000)
y = 10*np.random.normal(mu, sigma, 5000)
z = 10*np.random.normal(mu, sigma, 5000)
xyz = np.vstack([x,y,z])
kde = stats.gaussian_kde(xyz)
xmin, ymin, zmin = x.min(), y.min(), z.min()
xmax, ymax, zmax = x.max(), y.max(), z.max()
xi, yi, zi = np.mgrid[xmin:xmax:30j, ymin:ymax:30j, zmin:zmax:30j]
coords = np.vstack([item.ravel() for item in [xi, yi, zi]])
cores = multiprocessing.cpu_count()
pool = multiprocessing.Pool(processes=cores)
results = pool.map(calc_kde, np.array_split(coords.T, 2))
density = np.concatenate(results).reshape(xi.shape)
figure = mlab.figure('DensityPlot')
grid = mlab.pipeline.scalar_field(xi, yi, zi, density)
min = density.min()
max=density.max()
mlab.pipeline.volume(grid, vmin=min, vmax=min + .5*(max-min))
mlab.axes()
mlab.show()