我通过f(A(i,j))来赋值给A(i,j)。函数f()没有二进制输出,所以掩码操作无法帮助。双重循环迭代每个单元格是否是最优解?
高效评估NumPy数组中每个单元格的函数
我通过f(A(i,j))来赋值给A(i,j)。函数f()没有二进制输出,所以掩码操作无法帮助。双重循环迭代每个单元格是否是最优解?
1
import numpy as np
def f(x):
return x * x + 3 * x - 2 if x > 0 else x * 5 + 8
f = np.vectorize(f) # or use a different name if you want to keep the original f
result_array = f(A) # if A is your Numpy array
如果进行向量化,直接指定明确的输出类型可能会更好:
f = np.vectorize(f, otypes=[np.float])
7
vectorize函数说明中给出的警告:*vectorize函数主要是为了方便而提供的,而不是为了性能。其实现本质上是一个for循环。* 因此,这很可能根本不会加速进程。 - Gabrielvectorize函数如何确定返回类型,这可能会导致错误。frompyfunc速度稍快,但返回的是dtype对象数组。两者都只能处理标量,而不能处理行或列。 - hpauljnp.vectorize,就可以让我的代码速度提升约20倍。 - Suuuehgi我相信我已经找到了更好的解决方案。将函数更改为Python通用函数(请参见documentation),这样可以在幕后进行并行计算。
可以使用C编写自己定制的ufunc,这肯定更有效率,或者通过调用内置工厂方法np.frompyfunc。经过测试,这比np.vectorize更有效率:
f = lambda x, y: x * y
f_arr = np.frompyfunc(f, 2, 1)
vf = np.vectorize(f)
arr = np.linspace(0, 1, 10000)
%timeit f_arr(arr, arr) # 307ms
%timeit f_arr(arr, arr) # 450ms
我也测试了更大的样本,改进是成比例的。如需比较其他方法的性能,请参见此帖子
import numpy, time
def A(e):
return e * e
def timeit():
y = numpy.arange(1000000)
now = time.time()
numpy.array([A(x) for x in y.reshape(-1)]).reshape(y.shape)
print(time.time() - now)
now = time.time()
numpy.fromiter((A(x) for x in y.reshape(-1)), y.dtype).reshape(y.shape)
print(time.time() - now)
now = time.time()
numpy.square(y)
print(time.time() - now)
输出
>>> timeit()
1.162431240081787 # list comprehension and then building numpy array
1.0775556564331055 # from numpy.fromiter
0.002948284149169922 # using inbuilt function
在这里,您可以清楚地看到numpy.fromiter使用平方函数的用户,可以使用您选择的任何一个。如果您的函数依赖于数组的索引i,j,可以迭代数组大小,例如for ind in range(arr.size),使用numpy.unravel_index根据您的一维索引和数组形状获取i,j等numpy.unravel_index。
这个答案受到了我在其他问题上的回答的启发here
当二维数组(或多维数组)是C或F连续时,将函数映射到二维数组的任务实际上与将函数映射到一维数组的任务几乎相同-我们只需要以这种方式查看它,例如通过np.ravel(A,'K')。
例如,可能的解决方案已在此处here中讨论过。
然而,当二维数组的内存不连续时,情况会变得有点复杂,因为如果轴的处理顺序错误,就可能会避免可能的缓存未命中。
Numpy已经有一套机制来以最佳顺序处理轴。使用这个机制的一种可能方法是np.vectorize。然而,numpy关于np.vectorize的文档指出它"主要是为了方便而提供的,而不是为了性能"——一个慢的Python函数仍然是一个带有所有相关开销的慢函数!另一个问题是它巨大的内存消耗——例如,请参见这个SO-post。当人们想要拥有C函数的性能但又想使用numpy的机制时,一个好的解决方案是使用numba来创建ufuncs,例如:
# runtime generated C-function as ufunc
import numba as nb
@nb.vectorize(target="cpu")
def nb_vf(x):
return x+2*x*x+4*x*x*x
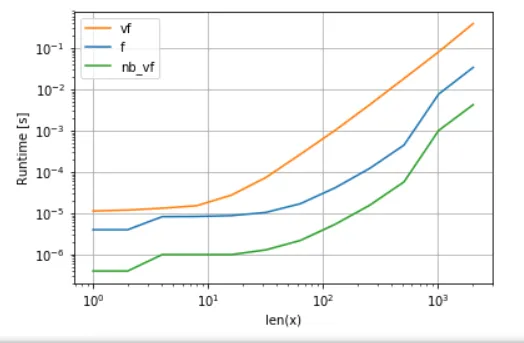
它轻松地击败了np.vectorize,而且当相同的函数作为numpy数组乘法/加法执行时也是如此。
# numpy-functionality
def f(x):
return x+2*x*x+4*x*x*x
# python-function as ufunc
import numpy as np
vf=np.vectorize(f)
vf.__name__="vf"
请参见本答案的附录以获取时间测量代码: Numba的版本(绿色)比Python函数(即
np.vectorize)快大约100倍,这并不奇怪。但它也比NumPy功能快约10倍,因为Numba的版本不需要中间数组,因此可以更有效地使用缓存。
虽然numba的ufunc方法在可用性和性能之间取得了良好的平衡,但仍不是我们能做到的最好的。然而,并没有银弹或适用于任何任务的最佳方法 - 人们必须了解限制以及如何缓解这些限制。
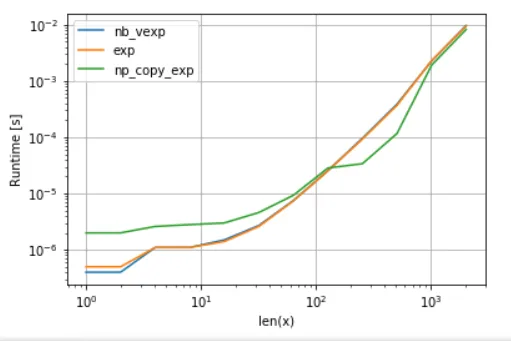
例如,对于超越函数(例如exp,sin,cos),numba与numpy的np.exp相比并没有提供任何优势(没有创建临时数组 - 这是加速的主要来源)。然而,我的Anaconda安装使用Intel的VML进行大于8192的向量bigger than 8192 - 如果内存不连续,则无法执行此操作。因此,最好将元素复制到连续的内存中,以便能够使用Intel的VML:
import numba as nb
@nb.vectorize(target="cpu")
def nb_vexp(x):
return np.exp(x)
def np_copy_exp(x):
copy = np.ravel(x, 'K')
return np.exp(copy).reshape(x.shape)
为了公平比较,我已经关闭了VML的并行化(请参见附录中的代码):
正如我们所看到的,一旦VML启动,复制的开销就会得到更好的补偿。然而,一旦数据变得太大,超出了L3缓存的范围,优势就变得微不足道,因为任务再次受到内存带宽的限制。
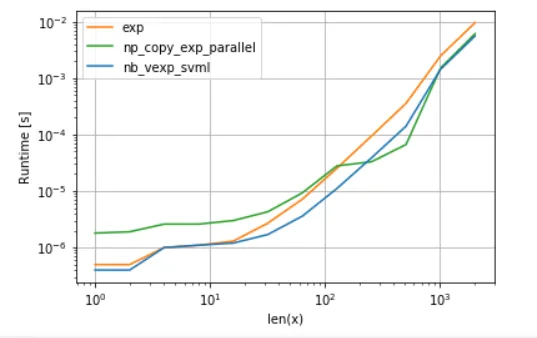
另一方面,numba也可以使用英特尔的SVML,就像this post中所解释的那样:
from llvmlite import binding
# set before import
binding.set_option('SVML', '-vector-library=SVML')
import numba as nb
@nb.vectorize(target="cpu")
def nb_vexp_svml(x):
return np.exp(x)
使用VML并行化的结果如下:
numba的版本开销较小,但对于某些大小的数据,VML甚至在额外的复制开销下也能击败SVML - 这并不奇怪,因为numba的ufuncs没有并行化。
列表:
A. 多项式函数的比较:
import perfplot
perfplot.show(
setup=lambda n: np.random.rand(n,n)[::2,::2],
n_range=[2**k for k in range(0,12)],
kernels=[
f,
vf,
nb_vf
],
logx=True,
logy=True,
xlabel='len(x)'
)
B. exp 函数的比较:
import perfplot
import numexpr as ne # using ne is the easiest way to set vml_num_threads
ne.set_vml_num_threads(1)
perfplot.show(
setup=lambda n: np.random.rand(n,n)[::2,::2],
n_range=[2**k for k in range(0,12)],
kernels=[
nb_vexp,
np.exp,
np_copy_exp,
],
logx=True,
logy=True,
xlabel='len(x)',
)
原文链接